k-means聚类方法和混合高斯模型
一、实验目的
-
目标
实现一个k-means算法和混合高斯模型,并且用EM算法估计模型中的参数。
-
测试
用高斯分布产生k个高斯分布的数据(不同均值和方差)(其中参数自己设定)。
(1)用k-means聚类,测试效果;
(2)用混合高斯模型和你实现的EM算法估计参数,看看每次迭代后似然值变化情况,考察EM算法是否可以获得正确的结果(与你设定的结果比较)。
-
应用
可以UCI上找一个简单问题数据,用你实现的GMM进行聚类。
二、实验环境
- win10
- python 3.7.4
- pycharm 2020.2
三、数学原理
-
EM
概率模型有时候即含有观察变量,又含有隐含变量。EM算法就是用来求解“含有隐变量的概率模型”的一种手段。
输入:观测变量数据 Y Y Y,隐含变量数据 Z Z Z,联合分布 P ( Y , Z ∣ θ ) P(Y,Z|theta) P(Y,Z∣θ),条件分布 P ( Z ∣ Y , θ ) P(Z|Y,theta) P(Z∣Y,θ)
输出:模型参数 θ theta θ
我们面对一个含有隐含变量的概率模型,目标是极大化观测数据(不完全数据)
Y
Y
Y关于参数
θ
theta
θ的对数似然函数,即极大化
L
(
θ
)
=
l
o
g
P
(
Y
∣
θ
)
=
l
o
g
∑
Z
P
(
Y
,
Z
∣
θ
)
=
l
o
g
(
∑
Z
P
(
Y
∣
Z
,
θ
)
P
(
Z
∣
θ
)
)
(1)
L(theta)=logP(Y|theta)=logsum_ZP(Y, Z|theta)=log(sum_ZP(Y|Z,theta)P(Z|theta)) tag{1}
L(θ)=logP(Y∣θ)=logZ∑P(Y,Z∣θ)=log(Z∑P(Y∣Z,θ)P(Z∣θ))(1)
但是这一极大化的主要困难是(1)式中有未观测数据并有包含和式的对数。
所以,我们转变思路,不求(1)式的解析解,而是通过迭代的方式逐步近似极大化 L ( θ ) L(theta) L(θ)。
假设在第
i
i
i次迭代之后
θ
theta
θ的估计值是
θ
(
i
)
theta^{(i)}
θ(i),我们希望新的估计值
θ
theta
θ能够使得
L
(
θ
)
>
L
(
θ
(
i
)
)
L(theta)>L(theta^{(i)})
L(θ)>L(θ(i)),并逐步到达最大值。为此,做差:
L
(
θ
)
−
L
(
θ
(
i
)
)
=
l
o
g
(
∑
Z
P
(
Y
∣
Z
,
θ
)
P
(
Z
∣
θ
)
)
−
l
o
g
P
(
Y
∣
θ
(
i
)
)
L(theta)-L(theta^{(i)})=log(sum_ZP(Y|Z,theta)P(Z|theta))-logP(Y|theta^{(i)})
L(θ)−L(θ(i))=log(Z∑P(Y∣Z,θ)P(Z∣θ))−logP(Y∣θ(i))
我们利用
J
E
N
S
E
N
JENSEN
JENSEN不等式得到上式的下界:
L
(
θ
)
−
L
(
θ
(
i
)
)
=
l
o
g
(
∑
Z
P
(
Z
∣
Y
,
θ
(
i
)
)
P
(
Y
∣
Z
,
θ
)
P
(
Z
∣
θ
)
P
(
Z
∣
Y
,
θ
(
i
)
)
)
−
l
o
g
P
(
Y
∣
θ
(
i
)
)
≥
∑
Z
P
(
Z
∣
Y
,
θ
(
i
)
)
l
o
g
P
(
Y
∣
Z
,
θ
)
P
(
Z
∣
θ
)
P
(
Z
∣
Y
,
θ
(
i
)
)
−
l
o
g
P
(
Y
∣
θ
(
i
)
)
=
∑
Z
P
(
Z
∣
Y
,
θ
(
i
)
)
l
o
g
P
(
Y
∣
Z
,
θ
)
P
(
Z
∣
θ
)
P
(
Z
∣
Y
,
θ
(
i
)
)
P
(
Y
∣
θ
(
i
)
)
begin{aligned} L(theta)-L(theta^{(i)})&=log(sum_ZP(Z|Y,theta^{(i)}) frac{P(Y|Z,theta)P(Z|theta)}{P(Z|Y,theta^{(i)})})-logP(Y|theta^{(i)})\ &gesum_ZP(Z|Y,theta^{(i)})logfrac{P(Y|Z,theta)P(Z|theta)}{P(Z|Y,theta^{(i)})}-logP(Y|theta^{(i)})\ &=sum_ZP(Z|Y,theta^{(i)})logfrac{P(Y|Z,theta)P(Z|theta)}{P(Z|Y,theta^{(i)})P(Y|theta^{(i)})} end{aligned}
L(θ)−L(θ(i))=log(Z∑P(Z∣Y,θ(i))P(Z∣Y,θ(i))P(Y∣Z,θ)P(Z∣θ))−logP(Y∣θ(i))≥Z∑P(Z∣Y,θ(i))logP(Z∣Y,θ(i))P(Y∣Z,θ)P(Z∣θ)−logP(Y∣θ(i))=Z∑P(Z∣Y,θ(i))logP(Z∣Y,θ(i))P(Y∣θ(i))P(Y∣Z,θ)P(Z∣θ)
令
B
(
θ
,
θ
(
i
)
)
=
L
(
θ
(
i
)
)
+
∑
Z
P
(
Z
∣
Y
,
θ
(
i
)
)
l
o
g
P
(
Y
∣
Z
,
θ
)
P
(
Z
∣
θ
)
P
(
Z
∣
Y
,
θ
(
i
)
)
P
(
Y
∣
θ
(
i
)
)
(2)
B(theta,theta^{(i)})=L(theta^{(i)})+sum_ZP(Z|Y,theta^{(i)})logfrac{P(Y|Z,theta)P(Z|theta)}{P(Z|Y,theta^{(i)})P(Y|theta^{(i)})} tag{2}
B(θ,θ(i))=L(θ(i))+Z∑P(Z∣Y,θ(i))logP(Z∣Y,θ(i))P(Y∣θ(i))P(Y∣Z,θ)P(Z∣θ)(2)
则
L
(
θ
)
≥
B
(
θ
,
θ
(
i
)
)
(3)
L(theta)ge B(theta,theta^{(i)}) tag{3}
L(θ)≥B(θ,θ(i))(3)
注意,上面用到的所谓
J
E
N
S
E
N
JENSEN
JENSEN不等式便是,对于一个凸函数,有
E
(
f
(
x
)
)
≥
f
(
E
(
x
)
)
E(f(x))ge f(E(x))
E(f(x))≥f(E(x))
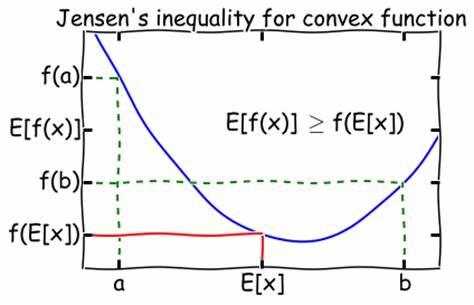
而
l
o
g
log
log函数是个凹函数,故不等号反向:
l
o
g
∑
j
λ
j
y
j
≥
∑
j
λ
j
l
o
g
y
j
logsum_j lambda_jy_j ge sum_j lambda_jlogy_j
logj∑λjyj≥j∑λjlogyj
其中
λ
j
≥
0
,
∑
j
λ
j
=
1
lambda_j ge 0, sum_j lambda_j=1
λj≥0,∑jλj=1
回到式(3),为了增大
L
(
θ
)
L(theta)
L(θ),我们选择
θ
(
i
+
1
)
theta^{(i+1)}
θ(i+1)使得
B
(
θ
,
θ
(
i
)
)
B(theta, theta^{(i)})
B(θ,θ(i))达到极大,即
θ
(
i
+
1
)
=
a
r
g
m
a
x
θ
B
(
θ
,
θ
(
i
)
)
(4)
theta^{(i+1)}=argmax_theta B(theta,theta^{(i)}) tag{4}
θ(i+1)=argmaxθB(θ,θ(i))(4)
现在求
θ
(
i
+
1
)
theta^{(i+1)}
θ(i+1)的表达式,省去常数项,有
θ
(
i
+
1
)
=
a
r
g
m
a
x
θ
(
L
(
θ
(
i
)
)
+
∑
Z
P
(
Z
∣
Y
,
θ
(
i
)
)
l
o
g
P
(
Y
∣
Z
,
θ
)
P
(
Z
∣
θ
)
P
(
Z
∣
Y
,
θ
(
i
)
)
P
(
Y
∣
θ
(
i
)
)
)
=
a
r
g
m
a
x
θ
(
∑
Z
P
(
Z
∣
Y
,
θ
(
i
)
)
l
o
g
(
P
(
Y
∣
Z
,
θ
)
P
(
Z
∣
θ
)
)
)
=
a
r
g
m
a
x
θ
(
∑
Z
P
(
Z
∣
Y
,
θ
(
i
)
)
l
o
g
P
(
Y
,
Z
∣
θ
)
=
a
r
g
m
a
x
θ
Q
(
θ
,
θ
(
i
)
)
(5)
begin{aligned} theta^{(i+1)}&=argmax_theta (L(theta^{(i)})+sum_ZP(Z|Y,theta^{(i)})logfrac{P(Y|Z,theta)P(Z|theta)}{P(Z|Y,theta^{(i)})P(Y|theta^{(i)})})\ &=argmax_theta(sum_ZP(Z|Y,theta^{(i)})log(P(Y|Z,theta)P(Z|theta)))\ &=argmax_theta(sum_ZP(Z|Y,theta^{(i)})logP(Y,Z|theta)\ &=argmax_theta Q(theta,theta^{(i)}) end{aligned} tag{5}
θ(i+1)=argmaxθ(L(θ(i))+Z∑P(Z∣Y,θ(i))logP(Z∣Y,θ(i))P(Y∣θ(i))P(Y∣Z,θ)P(Z∣θ))=argmaxθ(Z∑P(Z∣Y,θ(i))log(P(Y∣Z,θ)P(Z∣θ)))=argmaxθ(Z∑P(Z∣Y,θ(i))logP(Y,Z∣θ)=argmaxθQ(θ,θ(i))(5)
(5)式子等价于EM算法的一次迭代过程,即求
Q
Q
Q函数及其极大化。
总的来说,EM算法是通过不断求解下界的极大化来逼近求解对数似然函数极大值的算法。
直观解释如下:
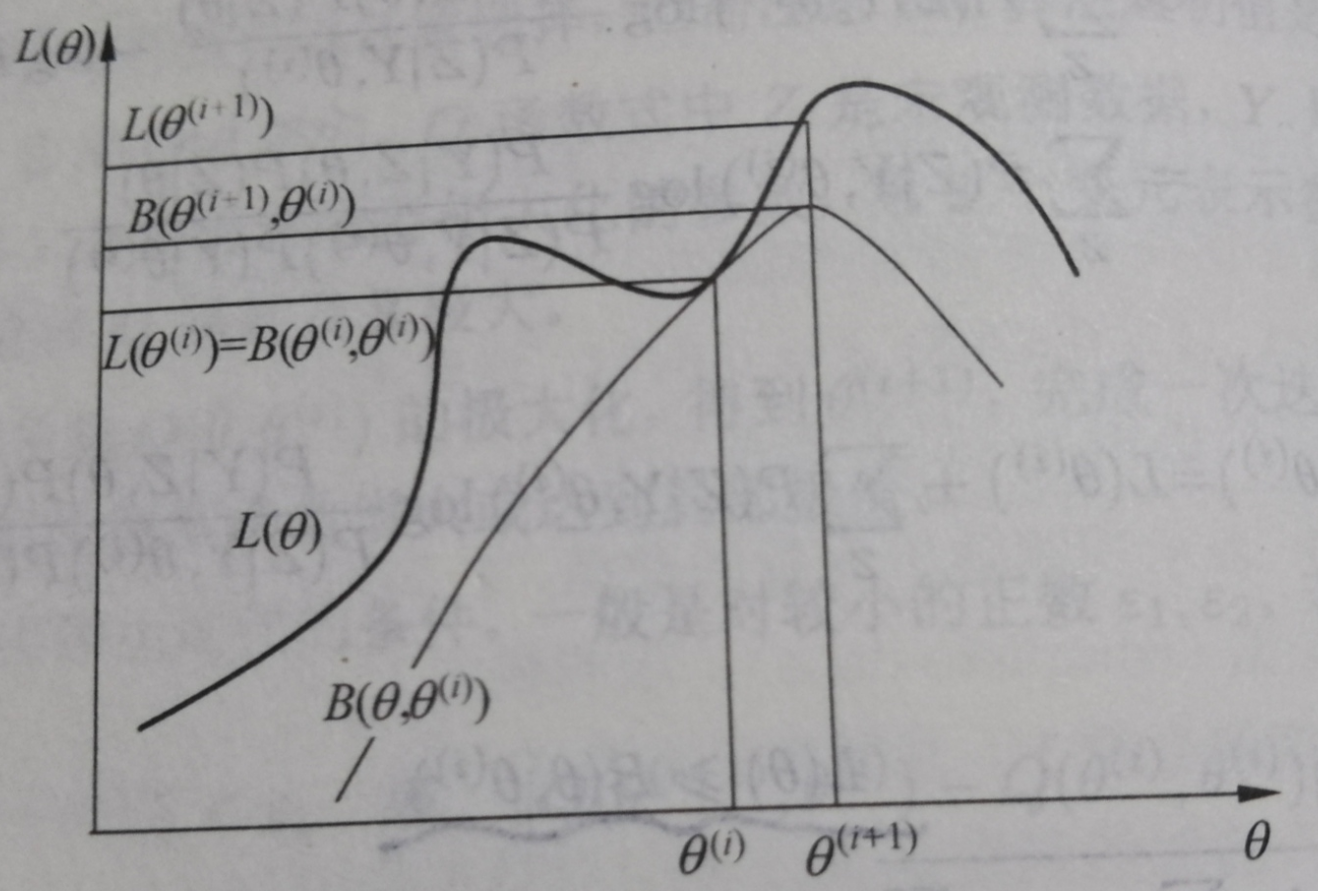
EM算法现在当前估计值 θ ( i ) theta^{(i)} θ(i)处找对数似然函数的下界 B ( θ , θ ( i + 1 ) ) B(theta,theta^{(i+1)}) B(θ,θ(i+1))使得其在 θ ( i ) theta^{(i)} θ(i)处的值和 L ( θ ) L(theta) L(θ)相同。再保持此下界不变,利用极大似然估计更新 θ ( i + 1 ) theta^{(i+1)} θ(i+1)。如此反复迭代,便可找到最佳 θ theta θ
下面对EM算法的步骤进行总结:
-
选择模型参数 θ theta θ的初始值 θ ( 0 ) theta^{(0)} θ(0)
-
E步:记 θ ( i ) theta^{(i)} θ(i)为第 i i i次迭代参数 θ theta θ的估计值,在第 i + 1 i+1 i+1次迭代的E步,找下界函数
Q ( θ , θ ( i ) ) = ∑ Z l o g ( P ( Y , Z ∣ θ ) P ( Z ∣ Y , θ ( i ) ) ) (6) Q(theta,theta^{(i)})=sum_Zlog(P(Y,Z|theta)P(Z|Y,theta^{(i)})) tag{6} Q(θ,θ(i))=Z∑log(P(Y,Z∣θ)P(Z∣Y,θ(i)))(6) -
M步:求使得下界函数 Q ( θ , θ ( i ) ) Q(theta,theta^{(i)}) Q(θ,θ(i))极大化的参数 θ theta θ,从而确定第 i + 1 i+1 i+1次迭代的参数估计值 θ ( i + 1 ) theta^{(i+1)} θ(i+1)
-
重复E步和M步,直到 θ theta θ收敛
值得注意的是,EM算法对初值是敏感的。
另外,也可证明出EM算法一定会收敛,且能找到局部最优解;若对数似然函数 L ( θ ) L(theta) L(θ)是凸函数或者凹函数,则能更进一步地确保找到全局最优解。
-
K-means
K-means聚类其实是EM算法一个应用实例,它用来将 N N N个数据点分为 K K K簇, K K K为事先给定。
首先,采用欧氏距离来衡量2个数据点之间的差异。其次,我们依据此定义样本与其所属簇中心点的距离的综合为损失函数,即
W
(
C
)
=
∑
l
=
1
k
∑
C
(
i
)
=
l
∣
∣
x
i
−
x
‾
l
∣
∣
2
(7)
W(C)=sum_{l=1}^ksum_{C(i)=l}||x_i-overline x_l||^2 tag{7}
W(C)=l=1∑kC(i)=l∑∣∣xi−xl∣∣2(7)
式子中
x
‾
l
overline x_l
xl是第
l
l
l簇的均值,
I
∗
=
(
C
(
i
)
)
=
l
I*=(C(i))=l
I∗=(C(i))=l是指示函数,取值为1或者0。
k-means聚类问题其实就是求解最优化问题
C
∗
=
a
r
g
m
a
x
C
∑
l
=
1
k
∑
C
(
i
)
=
l
∣
∣
x
i
−
x
‾
l
∣
∣
2
(8)
C^*=argmax_C sum_{l=1}^ksum_{C(i)=l}||x_i-overline x_l||^2 tag{8}
C∗=argmaxCl=1∑kC(i)=l∑∣∣xi−xl∣∣2(8)
倘若遍历所有分类方式,我们会发现这是个
N
P
NP
NP难问题,故我们利用迭代的方式来求解,也就是使用EM的思路。
输入: n n n个样本的集合 X X X,以及簇的数目 K K K
输出:样本集合的聚类 C ∗ C^* C∗
步骤:
- 初始化 K K K个中心点 ( m 1 ( 0 ) , m 2 ( 0 ) . . . m k ( 0 ) ) (m_1^{(0)},m_2^{(0)}...m_k^{(0)}) (m1(0),m2(0)...mk(0))
- 对样本点进行聚类。对固定的类中心 ( m 1 ( t ) , m 2 ( t ) . . . m k ( t ) ) (m_1^{(t)},m_2^{(t)}...m_k^{(t)}) (m1(t),m2(t)...mk(t)),计算每个样本到类中心的距离,将每个样本指派到与其最近的中心的类中,构成聚类结果 C ( t ) C^{(t)} C(t)
- 计算新的类中心,对聚类结果 C ( t ) C^{(t)} C(t),计算当前各个类中的样本的均值,作为新的类中心 ( m 1 ( t + 1 ) , m 2 ( t + 1 ) . . . m k ( t + 1 ) ) (m_1^{(t+1)},m_2^{(t+1)}...m_k^{(t+1)}) (m1(t+1),m2(t+1)...mk(t+1))
- 重复2和3步,直到类中心收敛
-
GMM
我们可以使用EM算法来处理高斯混合模型,也就是说,GMM同样是EM的一个应用实例。
所谓GMM,是指具有如下形式的概率分布模型:
P
(
y
∣
θ
)
=
∑
k
=
1
K
α
k
ϕ
(
y
∣
θ
k
)
(9)
P(y|theta)=sum_{k=1}^Kalpha_kphi(y|theta_k) tag{9}
P(y∣θ)=k=1∑Kαkϕ(y∣θk)(9)
其中
α
k
alpha_k
αk是系数,
α
k
≥
0
,
∑
k
=
1
K
α
k
=
1
alpha_k ge 0, sum_{k=1}^K alpha_k=1
αk≥0,∑k=1Kαk=1;
ϕ
(
y
∣
θ
k
)
phi(y|theta_k)
ϕ(y∣θk)是高斯密度函数,
θ
k
=
(
μ
k
,
σ
k
2
)
theta_k=(mu_k,sigma_k^2)
θk=(μk,σk2)
ϕ
(
y
∣
θ
k
)
=
1
2
π
σ
k
e
x
p
(
−
(
y
−
μ
k
)
2
2
σ
2
)
(10)
phi(y|theta_k)=frac{1}{sqrt{2pi}sigma_k}exp(-frac{(y-mu_k)^2}{2sigma^2}) tag{10}
ϕ(y∣θk)=2πσk1exp(−2σ2(y−μk)2)(10)
称为第
k
k
k个分模型。
现在假设观测数据
y
1
,
y
2
.
.
.
y
N
y_1,y_2...y_N
y1,y2...yN由高斯混合模型生成
P
(
y
∣
θ
)
=
∑
k
=
1
K
α
k
ϕ
(
y
∣
θ
k
)
P(y|theta)=sum_{k=1}^Kalpha_kphi(y|theta_k)
P(y∣θ)=k=1∑Kαkϕ(y∣θk)
其中
θ
=
(
α
1
,
α
2
.
.
.
α
k
;
θ
1
,
θ
2
.
.
.
θ
k
)
theta=(alpha_1,alpha_2...alpha_k;theta_1,theta_2...theta_k)
θ=(α1,α2...αk;θ1,θ2...θk)是模型参数,我们使用EM算法的思路来估计
θ
theta
θ
-
明确隐含变量,写出完全数据的对数似然函数
反映观测数据 y j y_j yj来自第 k k k个分模型的数据是未知的,我们以隐含变量 r j k r_{jk} rjk来表示 r j k = 1 r_{jk}=1 rjk=1表示 y j y_j yj来自第 k k k个分模型, r j k r_{jk} rjk是0-1随机变量。
那么完全数据的似然函数是
P
(
y
,
r
∣
θ
)
=
∏
j
=
1
N
P
(
y
j
,
r
j
1
,
r
j
2
.
.
.
r
j
k
∣
θ
)
P(y,r|theta)=prod_{j=1}^NP(y_j,r_{j1},r_{j2}...r_{jk}|theta)
P(y,r∣θ)=j=1∏NP(yj,rj1,rj2...rjk∣θ)
对数似然函数为
l
o
g
P
(
y
,
r
∣
θ
)
=
∑
k
=
1
K
(
n
k
l
o
g
α
k
+
∑
j
=
1
N
r
j
k
[
l
o
g
(
1
2
π
)
−
l
o
g
σ
k
−
1
2
σ
2
(
y
j
−
μ
k
)
2
)
]
)
(11)
logP(y,r|theta)=sum_{k=1}^K(n_klogalpha_k+sum_{j=1}^Nr_{jk}[log(frac{1}{sqrt{2pi}})-logsigma_k-frac{1}{2sigma^2}(y_j-mu_k)^2)]) tag{11}
logP(y,r∣θ)=k=1∑K(nklogαk+j=1∑Nrjk[log(2π1)−logσk−2σ21(yj−μk)2)])(11)
-
E步:确定Q函数
Q ( θ , θ ( i ) ) = E [ l o g P ( y , r ∣ θ ) ∣ y , θ ( i ) ] = ∑ k = 1 K ( n k l o g α k + ∑ j = 1 N r ^ j k [ l o g ( 1 2 π ) − l o g σ k − 1 2 σ 2 ( y j − μ k ) 2 ) ] ) begin{aligned} Q(theta,theta^{(i)})&=E[logP(y,r|theta)|y,theta^{(i)}]\ &=sum_{k=1}^K(n_klogalpha_k+sum_{j=1}^N hat r_{jk}[log(frac{1}{sqrt{2pi}})-logsigma_k-frac{1}{2sigma^2}(y_j-mu_k)^2)]) end{aligned} Q(θ,θ(i))=E[logP(y,r∣θ)∣y,θ(i)]=k=1∑K(nklogαk+j=1∑Nr^jk[log(2π1)−logσk−2σ21(yj−μk)2)])
其中
r
^
j
k
=
α
k
ϕ
(
y
j
∣
θ
k
)
∑
k
=
1
K
α
k
ϕ
(
y
j
∣
θ
k
)
hat r_{jk} = frac {alpha_kphi(y_j|theta_k)}{sum_{k=1}^Kalpha_kphi(y_j|theta_k)}
r^jk=∑k=1Kαkϕ(yj∣θk)αkϕ(yj∣θk)
称为分模型
k
k
k对观测数据
y
j
y_j
yj的响应度。
-
M步:极大似然法更新参数
μ k = ∑ j = 1 N r ^ j k y j ∑ j = 1 N r ^ j k mu_k =frac{sum_{j=1}^N hat r_{jk}y_j}{sum_{j=1}^N hat r_{jk}} μk=∑j=1Nr^jk∑j=1Nr^jkyj
σ k 2 = ∑ j = 1 N r ^ j k ( y j − μ k ) 2 ∑ j = 1 N r ^ j k sigma_k^2=frac{sum_{j=1}^N hat r_{jk}(y_j-mu_k)^2}{sum_{j=1}^Nhat r_{jk}} σk2=∑j=1Nr^jk∑j=1Nr^jk(yj−μk)2
α k = ∑ j = 1 N r ^ j k N alpha_k=frac{sum_{j=1}^N hat r_{jk}}{N} αk=N∑j=1Nr^jk
-
重复E步和M步,直到模型参数 θ theta θ收敛
四、实验过程
-
生成数据
为便于作图及观察,我自己生成的数据均为2维数据,且数据的分布均为2维高斯分布。
代码如下:
def generate_data(sample_means, sample_number, k):
""" 生成多簇二维高斯分布数据
:argument k k类
:argument sample_means k类数据的均值 如[[2, 4],[-2, -4], [3, -6]]
:argument sample_number k类数据的数量 如[100, 200, 300]
"""
# 协方差矩阵设置为对角阵,且2个维度的方差均为0.1
cov = [[0.1, 0], [0, 0.1]]
data = []
for index in range(k):
for times in range(sample_number[index]):
data.append(np.random.multivariate_normal(
[sample_means[index][0], sample_means[index][1]], cov).tolist())
return np.array(data)
我们在本实验中选择 K = 3 K=3 K=3,每个高斯分布的均值点分别为 ( 2 , 4 ) , ( 3 , − 6 ) , ( − 2 , − 4 ) (2,4),(3,-6),(-2,-4) (2,4),(3,−6),(−2,−4),且分别产生100、200、300个数据点,即一共500个数据点
k = 3
means = [[2, 4], [3, -6], [-2, -4]]
number = [100, 200, 300]
-
k-means聚类
所用算法的原理已经在前面给出,这里仅展示关键代码
# 用K-means求解,返回K个簇和它们的中心点
def Solve(self):
ROUND = 0
while True:
# 打印当前的迭代情况
print("K-means : ROUND " + str(ROUND))
print("The centers are : n", self.__mu)
ROUND += 1
c = collections.defaultdict(list)
# 对每个数据点进行聚类
for i in range(self.dataNumber):
# 求出每个数据点到各个中心点的距离
dij = [Bo.distance(self.data[i], self.__mu[j]) for j in range(self.k)]
# 将当前数据点归类到距离最近的那一簇中
labelIndex = np.argmin(dij)
c[labelIndex].append(self.data[i].tolist())
# 对每个中心点进行更新
new_mu = np.array([np.mean(c[i], axis=0).tolist() for i in range(self.k)])
# 检测loss是否达到精度要求
loss = np.sum(Bo.distance(self.__mu[i], new_mu[i]) for i in range(self.k))
if loss > self.delta:
self.__mu = new_mu
continue
else:
break
return self.__mu, c
同时,前面的数学原理部分,我们提到了EM算法对初始值敏感,因此我们在此处设置了对照试验:随机的初始点和优化的初始点
-
所谓随机的初始点,便是在500个数据点中随机抽取3个作为3个簇的中心点。
-
所谓优化的初始点,我这里采用了一种贪心的策略来尽可能保证各个簇的中心点彼此远离。
# 随机选第1个初始点
index = np.random.randint(0, self.k)
mu = [self.data[index]]
# 依次选择与当前mu中样本点距离之和最大的点作为初始簇中心点
for times in range(self.k-1):
distanceSum = []
for i in range(self.dataNumber):
distanceSum.append(np.sum([Bo.distance(self.data[i], mu[j]) for j in range(len(mu))]))
mu.append(self.data[np.argmax(distanceSum)])
产生的对比图如下:
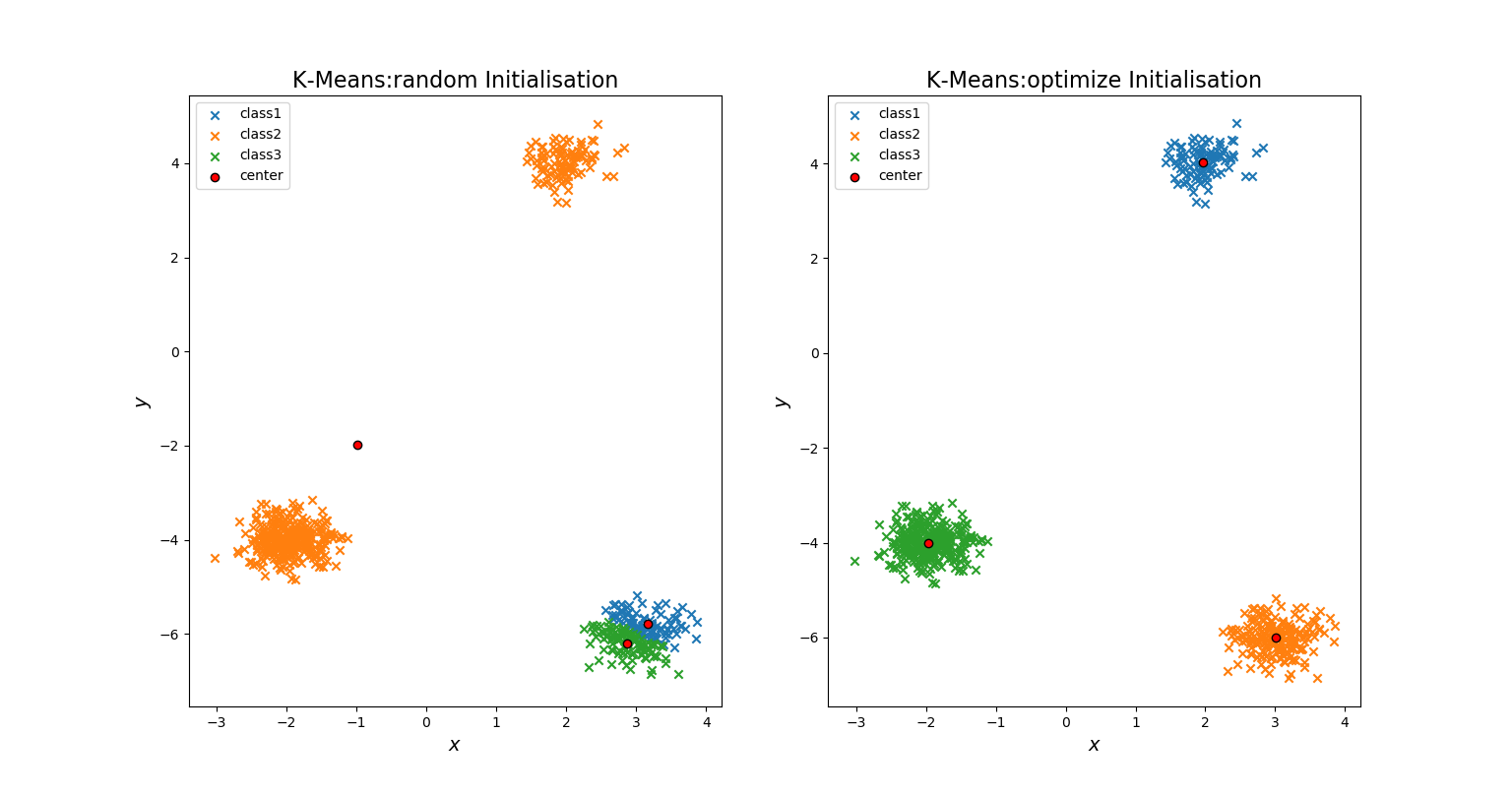
可以发现,“随机选择初始点”的聚类效果不如“优化选择初始点”的聚类效果好。
我们再将优化选择初始点的K-means算法求出的模型参数打印出来:

可以发现它求出的中心点和我们初始时设置的
means = [[2, 4], [3, -6], [-2, -4]]
极为接近。
此外,我寻找了一个衡量聚类效果好坏的指标:轮廓系数(Silhouette Coefficient)
是聚类效果好坏的一种评价方式。最早由 Peter J. Rousseeuw 在 1986 提出。它结合内聚度和分离度两种因素。可以用来在相同原始数据的基础上用来评价不同算法、或者算法不同运行方式对聚类结果所产生的影响。
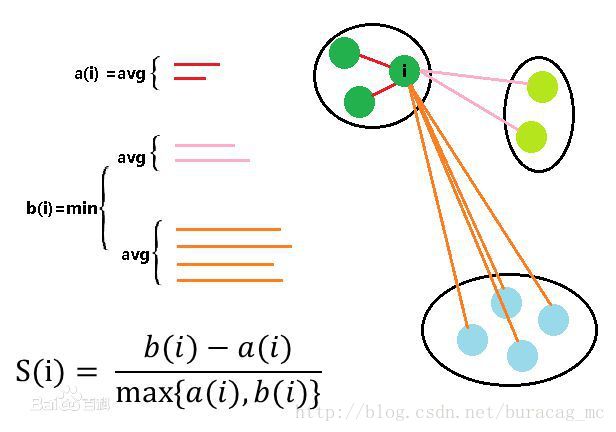
假设我们已经通过聚类算法将待分类数据进行了聚类,分为了 k 个簇 。对于簇中的每个向量。分别计算它们的轮廓系数。
对于其中的一个点 i 来说:
-
计算 a(i) = average(i向量到所有它属于的簇中其它点的距离)
-
计算 b(i) = min (i向量到所有非本身所在簇的点的平均距离)
那么 i 向量轮廓系数就为:
S ( i ) = b ( i ) − a ( i ) m a x ( a ( i ) , b ( i ) ) S(i)=frac{b(i)-a(i)}{max(a(i),b(i))} S(i)=max(a(i),b(i))b(i)−a(i)
最后将所有样本点的轮廓系数求平均,就是该聚类结果总的轮廓系数。
可见轮廓系数的值是介于 [-1,1] ,越趋近于1代表内聚度和分离度都相对较优。
下面我们计算出K-means聚类结果的轮廓系数

可见,我们的优化初始点的K-means聚类效果还是很不错的。
-
GMM聚类
首先生成数据,代码如下:
def GMMData():
# 第一簇的数据
num1, mu1, var1 = 200, [0.5, 0.5], [1, 3]
X1 = np.random.multivariate_normal(mu1, np.diag(var1), num1)
# 第二簇的数据
num2, mu2, var2 = 300, [5.5, 2.5], [2, 2]
X2 = np.random.multivariate_normal(mu2, np.diag(var2), num2)
# 第三簇的数据
num3, mu3, var3 = 500, [1, 7], [6, 2]
X3 = np.random.multivariate_normal(mu3, np.diag(var3), num3)
# 合并在一起
X = np.vstack((X1, X2, X3))
true_mu = [mu1, mu2, mu3]
true_Var = [var1, var2, var3]
return X, X1, X2, X3, true_mu, true_Var
产生的数据图如下:
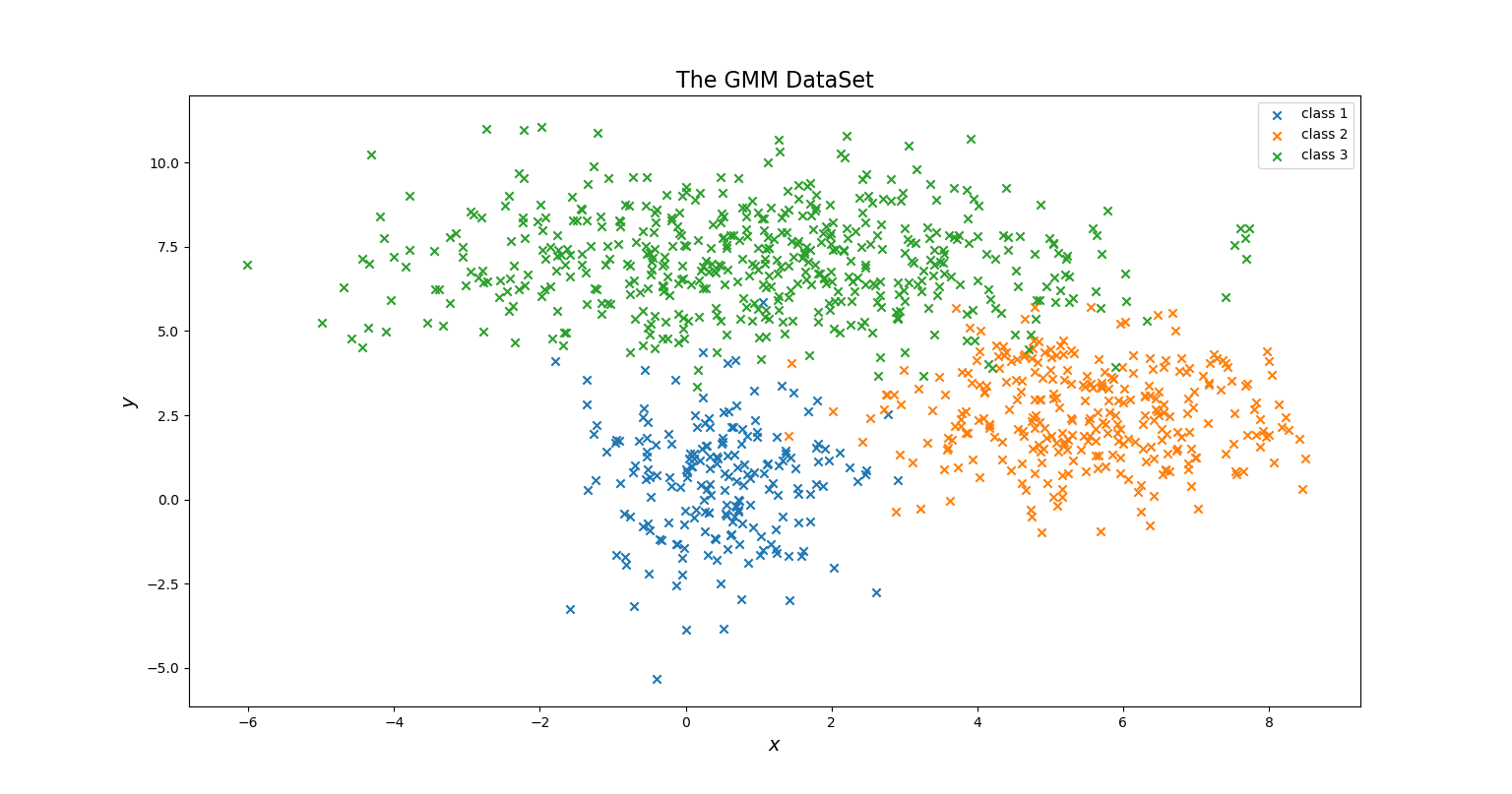
下面开始聚类,所用算法的原理已经在前面给出,这里仅展示关键代码
# E步
def E_step(self):
# 计算概率密度和alpha之积
weighted = self.probability_density() * self.__alpha
sum_likelihoods = np.expand_dims(np.sum(weighted, axis=1), axis=1)
# 返回当前的似然值
value = np.log(np.prod(sum_likelihoods))
# 求出gamma,即分模型对观测数据的“响应度矩阵” (N*K)
self.__gamma = weighted / sum_likelihoods
self.sample_assignments = self.__gamma.argmax(axis=1)
# 依据响应度将每个数据点进行聚类
for i in range(self.dataNumber):
self.c[self.sample_assignments[i]].append(self.data[i].tolist())
return value
# M步
def M_step(self):
# 在隐参数保持不变的情况下使用极大似然法更新模型参数
for i in range(self.k):
# 提取每一列 作为列向量 (m, 1)
gamma = np.expand_dims(self.__gamma[:, i], axis=1)
mean = (gamma * self.data).sum(axis=0) / gamma.sum()
covariance = (self.data - mean).T.dot((self.data - mean) * gamma) / gamma.sum()
# 更新均值和方差
self.__mu[i], self.__sigma[i] = mean, covariance
# 更新各个分布的权值
self.__alpha = self.__gamma.sum(axis=0) / self.dataNumber
至于初始化的问题,由于前面已经验证了“优化选择初始点”的优越性,我们这里继续沿用。
下面展示聚类图像
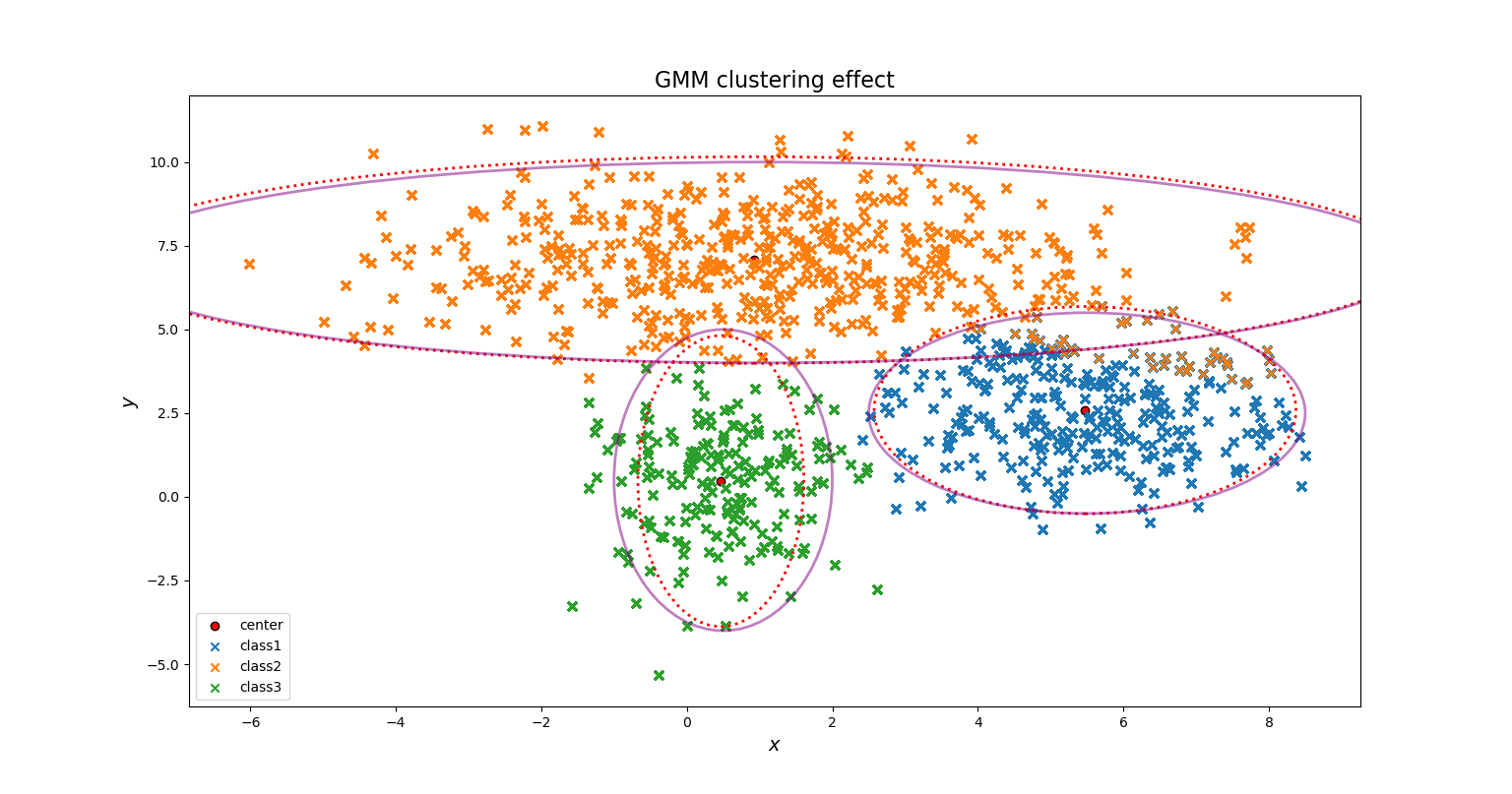
直观上看,我们的GMM聚类效果还是不错的。
至于图中的椭圆,其实是依据各个高斯分布的概率密度而做出的边界,红色虚线表示用于真正生成数据集的“高斯分布椭圆”,紫色实线表示通过GMM学习到的“高斯分布椭圆”
因为对于一个高斯分布而言,它的数据点几乎全部分布在 ( μ − 3 σ , μ + 3 σ ) (mu-3sigma, mu+3sigma) (μ−3σ,μ+3σ)之间,因此我们常常用这一区间的边界图形来表示一个高斯分布。而对于一个二维的高斯分布而言,它的边界就是个椭圆。
下面我们打印出GMM求出的模型参数
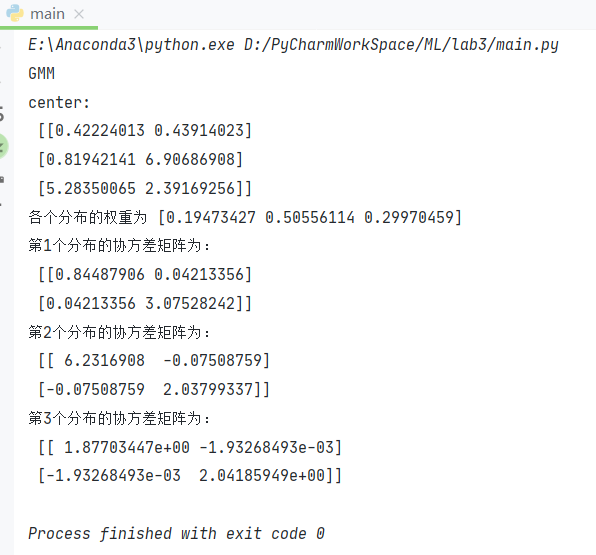
可见与我们设定的值
means = [0.5,0.5],[1,7],[5.5,2.5]
sigma = [np.diag([1,3]),np.diag([6,2]),np.diag([2,2])]
alpha = [0.2,0.3,0.5]
是十分接近的。
我们再计算它的轮廓系数:

因为轮廓系数已经很接近1了,可见GMM聚类算法的效果很不错。
最后,我们将整个迭代过程的似然值变化图线画出来:
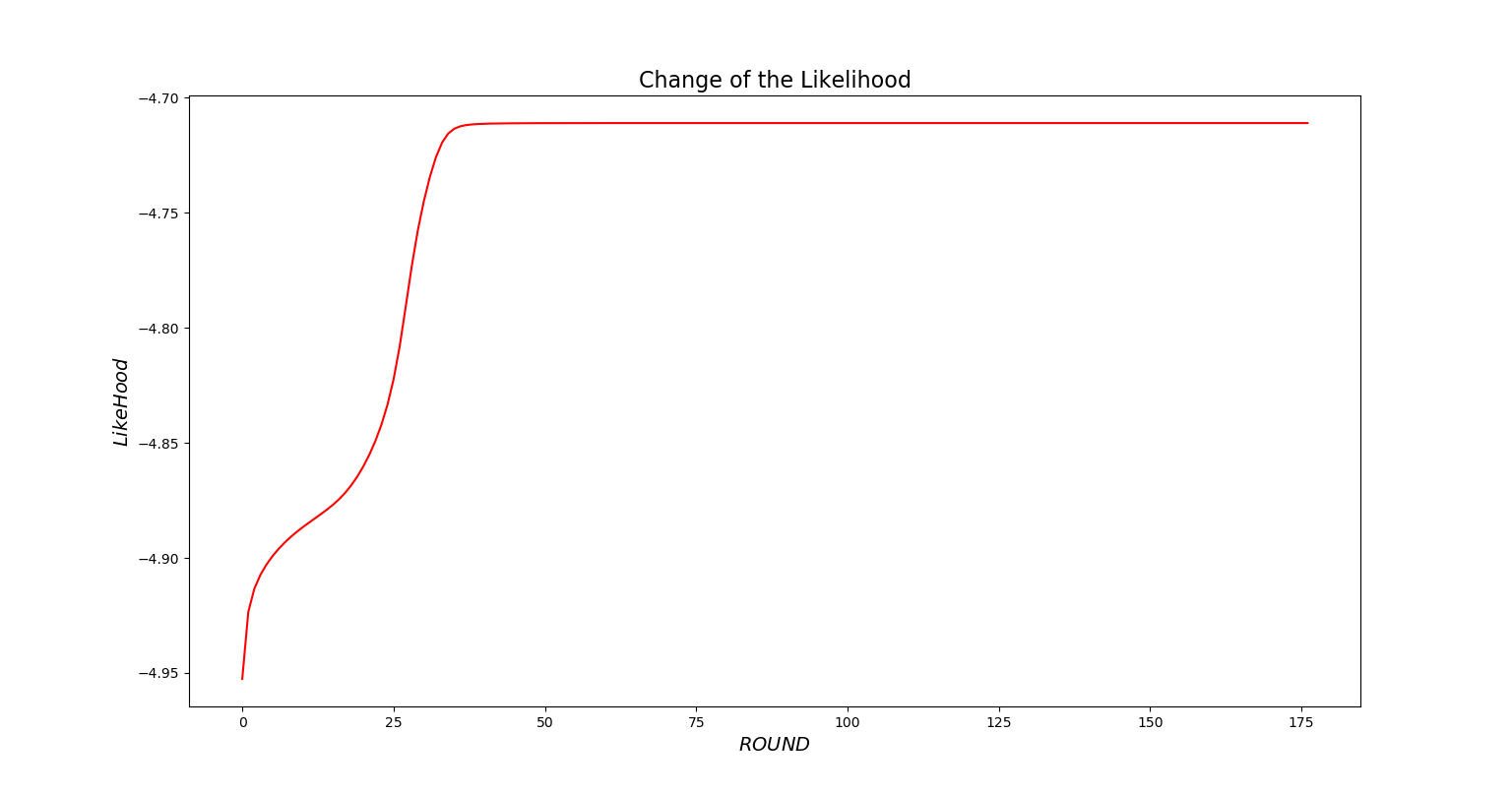
可见,随着迭代轮数的增加,似然值在不断增大,最终收敛。
-
UCI数据集测试
我们使用IRIS数据集对我们的GMM算法进行测试
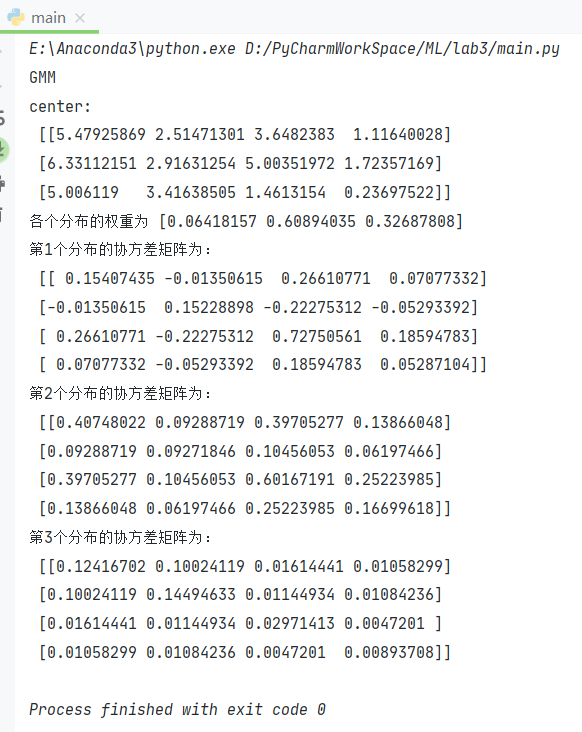
下面展示使用GMM聚类后得到的模型参数
展示轮廓系数

可见我们的聚类效果还不错。
展示似然值随着迭代轮数的变化
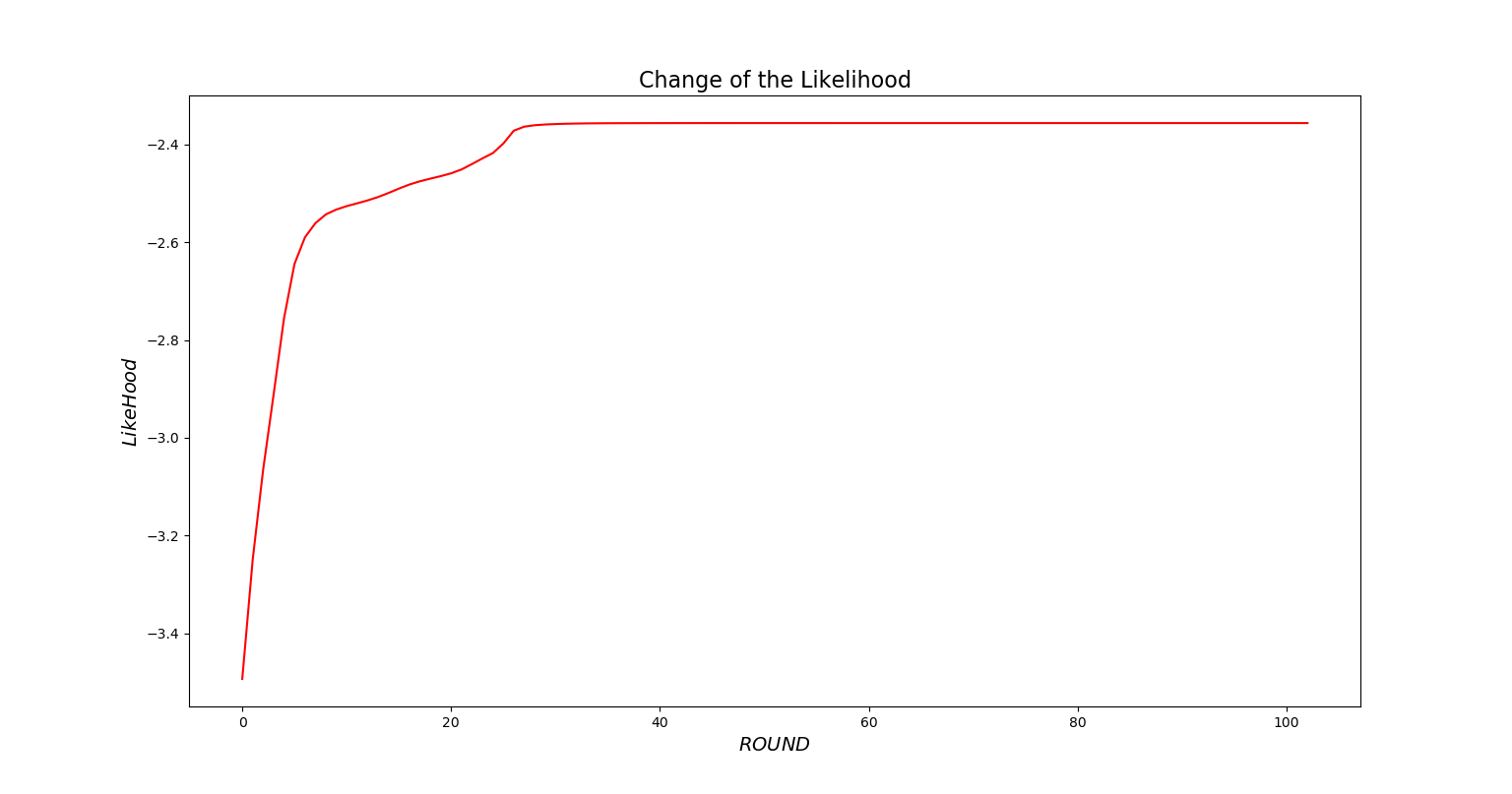
五、实验结论
- EM算法对初始值敏感,若初始值选取不好,聚类的效果会差很多
- K-means和GMM是EM的2个具体应用实例
- K-Means采用欧式距离衡量数据点的差异,故它假设数据呈球状分布,每个维度的重要性一样;与之相比GMM使用更加一般的数据表示即高斯分布,它考虑到了协方差
六、源代码
main.py
import k_means
import basicOperation as Bo
import drawImage as dI
import numpy as np
import gmm
k = 3
means = [[2, 4], [3, -6], [-2, -4]]
number = [100, 200, 300]
data = Bo.generate_data(means, number, k)
# 使用K-means
km = k_means.KMeans(data, k)
mu_random, c_random = km.RandomCenterAnswer()
# print("随机初始点的轮廓系数为", Bo.SilhouetteCoefficient(c_random))
print("-----------------")
mu_NotRandom, c_NotRandom = km.NotRandomCenterAnswer()
# print("优化初始但的轮廓系数为", Bo.SilhouetteCoefficient(c_NotRandom))
dI.KMeansImage(k, mu_random, c_random, mu_NotRandom, c_NotRandom)
data, X1, X2, X3, true_mu, true_Var = Bo.GMMData()
dI.ShowGMMData(X1, X2, X3)
# 使用GMM
GMM = gmm.GaussianMixtureModel(data, k=k)
# 前3个为模型参数,第四个为聚类的簇结果,第五个数迭代轮数的List,第六个是似然值List
alpha, mu, sigma, c, ROUNDList, likelihoodList = GMM.solve()
print("center: n", mu)
print("各个分布的权重为", alpha)
Bo.printSigma(sigma)
# 既将sigma从字典转为list, 又将协方差矩阵的多余cov去除
sigma = Bo.ChangeVar(sigma)
dI.GMMImage(c, mu, sigma, true_mu, true_Var)
print("GMM的轮廓系数为", Bo.SilhouetteCoefficient(c))
dI.LikeHoodChange(np.array(ROUNDList), np.array(likelihoodList))
iris_data = Bo.ReadUCI("UCIdata/iris.csv")
GMM = gmm.GaussianMixtureModel(iris_data, k=3)
alpha, mu, sigma, c, ROUNDList, likelihoodList = GMM.solve()
print("center: n", mu)
print("各个分布的权重为", alpha)
Bo.printSigma(sigma)
# print("对IRIS数据集使用GMM的轮廓系数为", Bo.SilhouetteCoefficient(c))
# 展示似然值随着迭代轮数的变化
dI.LikeHoodChange(np.array(ROUNDList), np.array(likelihoodList))
k-means.py
import numpy as np
import collections
import random
import basicOperation as Bo
class KMeans(object):
def __init__(self, data, k, delta=1e-6):
self.data = data
self.k = k
self.delta = delta
self.dataNumber, self.dimNumber = data.shape
self.__mu = []
# self.sample_assignments = [-1] * self.dataNumber
def RandomCenterAnswer(self):
""" 随机选择k个顶点作为初始簇中心点 """
self.__mu = self.data[random.sample(range(self.dataNumber), self.k)]
return self.Solve()
def NotRandomCenterAnswer(self):
""" 随机选择第一个簇中心点 再选出下一个和已有中心点距离之和最大的点作为新中心点 这样不断循环便可找到K个初始点 """
# 随机选第1个初始点
index = np.random.randint(0, self.k)
mu = [self.data[index]]
# 依次选择与当前mu中样本点距离之和最大的点作为初始簇中心点
for times in range(self.k-1):
distanceSum = []
for i in range(self.dataNumber):
distanceSum.append(np.sum([Bo.distance(self.data[i], mu[j]) for j in range(len(mu))]))
mu.append(self.data[np.argmax(distanceSum)])
self.__mu = np.array(mu)
return self.Solve()
# 用K-means求解,返回K个簇和它们的中心点
def Solve(self):
ROUND = 0
while True:
# 打印当前的迭代情况
print("K-means : ROUND " + str(ROUND))
print("The centers are : n", self.__mu)
ROUND += 1
c = collections.defaultdict(list)
# 对每个数据点进行聚类
for i in range(self.dataNumber):
# 求出每个数据点到各个中心点的距离
dij = [Bo.distance(self.data[i], self.__mu[j]) for j in range(self.k)]
# 将当前数据点归类到距离最近的那一簇中
labelIndex = np.argmin(dij)
c[labelIndex].append(self.data[i].tolist())
# self.sample_assignments[i] = labelIndex
# 对每个中心点进行更新
new_mu = np.array([np.mean(c[i], axis=0).tolist() for i in range(self.k)])
# 检测loss是否达到精度要求
loss = np.sum(Bo.distance(self.__mu[i], new_mu[i]) for i in range(self.k))
if loss > self.delta:
self.__mu = new_mu
continue
else:
break
return self.__mu, c
gmm.py
import numpy as np
from scipy.stats import multivariate_normal
import collections
import random
import basicOperation as Bo
class GaussianMixtureModel(object):
""" 高斯混合聚类EM算法 """
def __init__(self, data, k=3, delta=1e-12, max_iteration=1000):
self.data = data
self.k = k
self.delta = delta
self.max_iteration = max_iteration
self.dataNumber, self.dimNumber = self.data.shape
# 初始化各个分布的均值、协方差矩阵、各个分布的权值
self.__mu, self.__sigma, self.__alpha = self.InitParams()
# 一个N*K矩阵,代表第j个数据点来自第k个分布的概率,是隐参数
self.__gamma = None
self.sample_assignments = None
self.c = collections.defaultdict(list)
def InitParams(self):
# mu = np.array(self.data[random.sample(range(self.dataNumber), self.k)])
# 随机选择k个点作为初始点 极易陷入局部最小值
# 初始化均值
index = np.random.randint(0, self.k)
mu = [self.data[index]]
# 依次选择与当前mu中样本点距离之和最大的点作为初始簇中心点
for times in range(self.k - 1):
distanceSum = []
for i in range(self.dataNumber):
distanceSum.append(np.sum([Bo.distance(self.data[i], mu[j]) for j in range(len(mu))]))
mu.append(self.data[np.argmax(distanceSum)])
# 初始化每个分布的协方差矩阵
sigma = collections.defaultdict(list)
for i in range(self.k):
# 每个分布的协方差矩阵都默认为对角阵,且每个维度的方差默认为0.1
sigma[i] = np.eye(self.dimNumber, dtype=float) * 0.1
# 初始化每个分布的权值
alpha = np.ones(self.k) * (1.0 / self.k)
return mu, sigma, alpha
# 计算似然值
def logLH(self):
data = self.data
alpha = self.__alpha
Mu = self.__mu
Var = self.__sigma
n_points, n_clusters = len(data), len(alpha)
pdfs = np.zeros((n_points, n_clusters))
for i in range(n_clusters):
pdfs[:, i] = alpha[i] * multivariate_normal.pdf(data, Mu[i], np.diag(Var[i]))
return np.mean(np.log(pdfs.sum(axis=1)))
# 求出概率密度矩阵(N,K)
def probability_density(self):
likelihoods = np.zeros((self.dataNumber, self.k))
for i in range(self.k):
# pdf为概率密度函数
likelihoods[:, i] = multivariate_normal.pdf(self.data, self.__mu[i], self.__sigma[i])
return likelihoods
# E步
def E_step(self):
# 计算概率密度和alpha之积
weighted = self.probability_density() * self.__alpha
sum_likelihoods = np.expand_dims(np.sum(weighted, axis=1), axis=1)
# 求出gamma,即分模型对观测数据的“响应度矩阵” (N*K)
self.__gamma = weighted / sum_likelihoods
self.sample_assignments = self.__gamma.argmax(axis=1)
# 依据响应度将每个数据点进行聚类
for i in range(self.dataNumber):
self.c[self.sample_assignments[i]].append(self.data[i].tolist())
# M步
def M_step(self):
# 在隐参数保持不变的情况下使用极大似然法更新模型参数
for i in range(self.k):
# 提取每一列 作为列向量 (m, 1)
gamma = np.expand_dims(self.__gamma[:, i], axis=1)
mean = (gamma * self.data).sum(axis=0) / gamma.sum()
covariance = (self.data - mean).T.dot((self.data - mean) * gamma) / gamma.sum()
# 更新均值和方差
self.__mu[i], self.__sigma[i] = mean, covariance
# 更新各个分布的权值
self.__alpha = self.__gamma.sum(axis=0) / self.dataNumber
def solve(self):
print("GMM")
ROUNDList = []
likelihoodList = []
old_alpha = self.__alpha
old_mu = self.__mu
old_sigma = self.__sigma
# 不断迭代,以求解
for i in range(self.max_iteration):
# 依次执行E和M步
self.E_step()
self.M_step()
# 判断是否收敛
diff = np.linalg.norm(old_alpha - self.__alpha)
+ np.linalg.norm(np.array(old_mu) - np.array(self.__mu))
+ np.sum([np.linalg.norm(np.array(old_sigma[i]) - np.array(self.__sigma[i])) for i in range(self.k)])
# print("diff: ", diff)
ROUNDList.append(i)
# 就算每步迭代的似然值
likelihoodList.append(self.logLH())
# 未达到精度要求,则更新模型参数
if diff > self.delta:
old_alpha = self.__alpha
old_mu = self.__mu
old_sigma = self.__sigma
continue
else:
break
return self.__alpha, np.array(self.__mu), self.__sigma, self.c, ROUNDList, likelihoodList
basicOperation.py
import numpy as np
import pandas as pd
def generate_data(sample_means, sample_number, k):
""" 生成多簇二维高斯分布数据
:argument k k类
:argument sample_means k类数据的均值 如[[2, 4],[-2, -4], [3, -6]]
:argument sample_number k类数据的数量 如[100, 200, 300]
"""
# 协方差矩阵设置为对角阵,且2个维度的方差均为0.1
cov = [[0.1, 0], [0, 0.1]]
data = []
for index in range(k):
for times in range(sample_number[index]):
data.append(np.random.multivariate_normal(
[sample_means[index][0], sample_means[index][1]], cov).tolist())
return np.array(data)
# 求2点之间欧氏距离(二范数)
def distance(x1, x2):
return np.linalg.norm(x1 - x2)
def SilhouetteCoefficient(c):
N = 0
SList = []
for i in range(len(c)):
N += len(np.array(c[i]))
for j in range(len(np.array(c[i]))):
# 遍历每个点
point = np.array(c[i])[j]
a = 0
for k in range(len(np.array(c[i]))):
# 遍历其他同簇点
otherPoint = np.array(c[i])[k]
a += distance(point, otherPoint) / len(np.array(c[i]))
# 计算b
b = []
for m in range(len(c)):
# 找不同簇
if m != i:
tmp = 0
for n in range(len(np.array(c[m]))):
otherPoint = np.array(c[m])[n]
tmp += distance(point, otherPoint) / len(np.array(c[m]))
b.append(tmp)
b = np.array(b).min()
s = (b - a) / max(a, b)
SList.append(s)
result = np.array(SList).mean()
return result
def ReadUCI(path):
data_set = pd.read_csv(path) # linux 相对路径
x = data_set.drop('label', axis=1)
dataX = np.array(x, dtype=float)
return dataX
def GMMData():
# 第一簇的数据
num1, mu1, var1 = 200, [0.5, 0.5], [1, 3]
X1 = np.random.multivariate_normal(mu1, np.diag(var1), num1)
# 第二簇的数据
num2, mu2, var2 = 300, [5.5, 2.5], [2, 2]
X2 = np.random.multivariate_normal(mu2, np.diag(var2), num2)
# 第三簇的数据
num3, mu3, var3 = 500, [1, 7], [6, 2]
X3 = np.random.multivariate_normal(mu3, np.diag(var3), num3)
# 合并在一起
X = np.vstack((X1, X2, X3))
true_mu = [mu1, mu2, mu3]
true_Var = [var1, var2, var3]
return X, X1, X2, X3, true_mu, true_Var
def printSigma(sigma):
for i in range(len(sigma)):
array = sigma[i]
print("第"+str(i+1)+"个分布的协方差矩阵为:n", array)
def ChangeVar(sigma):
result = []
for i in range(len(sigma)):
cov = []
array = sigma[i]
num1 = array[0][0]
num2 = array[1][1]
cov.append(num1)
cov.append(num2)
result.append(cov)
return result
drawImage.py
import matplotlib.pyplot as plt
import numpy as np
from matplotlib.patches import Ellipse
def ShowGMMData(X1, X2, X3):
fig, ax = plt.subplots()
ax.scatter(X1[:, 0], X1[:, 1], marker="x", label="class 1")
ax.scatter(X2[:, 0], X2[:, 1], marker="x", label="class 2")
ax.scatter(X3[:, 0], X3[:, 1], marker="x", label="class 3")
ax.set_xlabel("$x$", fontsize=14)
ax.set_ylabel("$y$", fontsize=14)
ax.set_title("The GMM DataSet", fontsize=16)
ax.legend()
plt.show()
def KMeansImage(k, mu_random, c_random, mu_NotRandom, c_NotRandom):
fig, axes = plt.subplots(1, 2)
axes[0].set_title("K-Means:random Initialisation", fontsize=16)
for i in range(k):
axes[0].scatter(np.array(c_random[i])[:, 0], np.array(c_random[i])[:, 1], marker="x", label="class"+str(i + 1))
axes[0].scatter(mu_random[:, 0], mu_random[:, 1], facecolor="red", edgecolor="black", label="center")
axes[0].set_xlabel("$x$", fontsize=14)
axes[0].set_ylabel("$y$", fontsize=14)
axes[0].legend()
axes[1].set_title("K-Means:optimize Initialisation", fontsize=16)
for i in range(k):
axes[1].scatter(np.array(c_NotRandom[i])[:, 0], np.array(c_NotRandom[i])[:, 1], marker="x", label="class"+str(i + 1))
axes[1].scatter(mu_NotRandom[:, 0], mu_NotRandom[:, 1], facecolor="red", edgecolor="black", label="center")
axes[1].set_xlabel("$x$", fontsize=14)
axes[1].set_ylabel("$y$", fontsize=14)
axes[1].legend()
plt.show()
def GMMImage(c, Mu, Var, Mu_true=None, Var_true=None):
n_clusters = len(Mu)
ax = plt.gca()
# 画中心点
ax.scatter(Mu[:, 0], Mu[:, 1], facecolor="red", edgecolor="black", label="center")
# 画数据点
for i in range(n_clusters):
ax.scatter(np.array(c[i])[:, 0], np.array(c[i])[:, 1], marker="x", label="class" + str(i + 1))
# 画GMM学习出的高斯椭圆
for i in range(n_clusters):
plot_args = {'fc': 'None', 'lw': 2, 'ls': ':', 'edgecolor': "red"}
ellipse = Ellipse(Mu[i], 3 * Var[i][0], 3 * Var[i][1], **plot_args)
ax.add_patch(ellipse)
# 画真实的高斯椭圆
if (Mu_true is not None) & (Var_true is not None):
for i in range(n_clusters):
plot_args = {'fc': 'None', 'lw': 2, 'alpha': 0.5, 'edgecolor': "purple"}
ellipse = Ellipse(Mu_true[i], 3 * Var_true[i][0], 3 * Var_true[i][1], **plot_args)
ax.add_patch(ellipse)
ax.set_title("GMM clustering effect", fontsize=16)
ax.set_xlabel("$x$", fontsize=14)
ax.set_ylabel("$y$", fontsize=14)
ax.legend()
plt.show()
def LikeHoodChange(ROUNDList, likelihoodList):
fig, axes = plt.subplots()
axes.set_title("Change of the Likelihood", fontsize=16)
axes.plot(ROUNDList, likelihoodList, color="red")
axes.set_xlabel("$ROUND$", fontsize=14)
axes.set_ylabel("$LikeHood$", fontsize=14)
plt.show()
最后
以上就是彪壮酒窝最近收集整理的关于哈工大-机器学习-实验三:EM、k-means、GMMk-means聚类方法和混合高斯模型的全部内容,更多相关哈工大-机器学习-实验三内容请搜索靠谱客的其他文章。



![[kuangbin]数学训练四 数论 [Cloned]](https://www.shuijiaxian.com/files_image/reation/bcimg10.png)




发表评论 取消回复