叶绿体基因组PGA注释
PGA中文文档
测试数据依旧选择上两篇中用到的数据
Fasta文件为:MZ489116.1
Genbank文件为 NC_063470.1
PGA 程序运行:
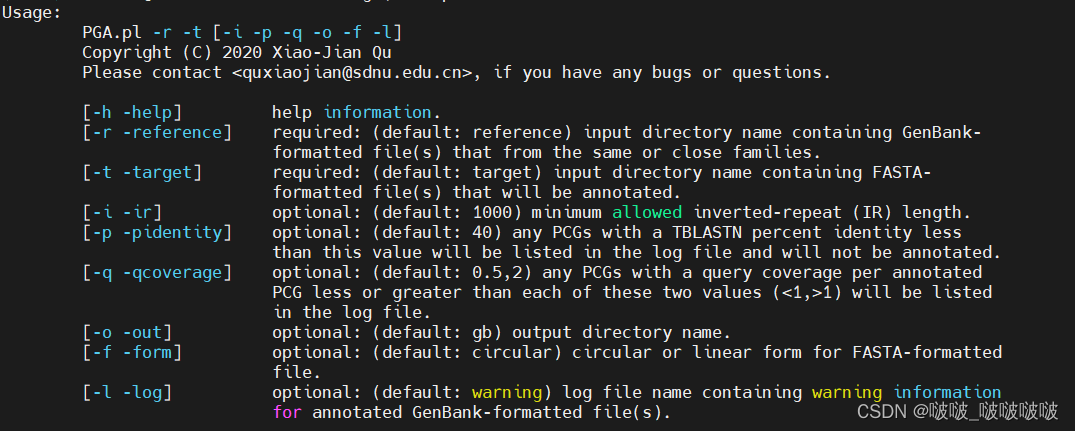
将PGA.pl 写入环境变量后运行
PGA.pl -r ./reference -t ./target -o output
运行成功之后会生成三个文件test.gb、warning.log、screen.log
查看warning.log 里面显示psbL 起始密码子出错,trnH-GUG 没有注释出来
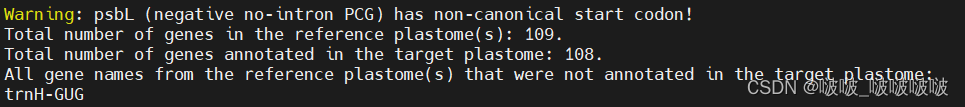
然后用Geseq对组装结果进行检查:


一共有四种错误:
psbL、rps19的起始密码子出错,rps12注释出错、clpP基因内部含有终止密码子。
psbL、rps12可以参照上一篇博客写的方法进行修改。内部含有终止密码子的clpP的修改则需要根据参考确定三个外显子的起始位置,然后进行合并。
而对于没有注释出来的基因trnH-GUG,则可以选择使用blastn进行比对
将参考中的trnH-GUG基因的序列提取出来,保存为trnH-GUG.fa 然后运行
blastn -query trnH-GUG.fa -subject test.fa -outfmt 6
然后按照参考进行修改,写入gb文件里面即可。
PGA结果修改成NCBI格式
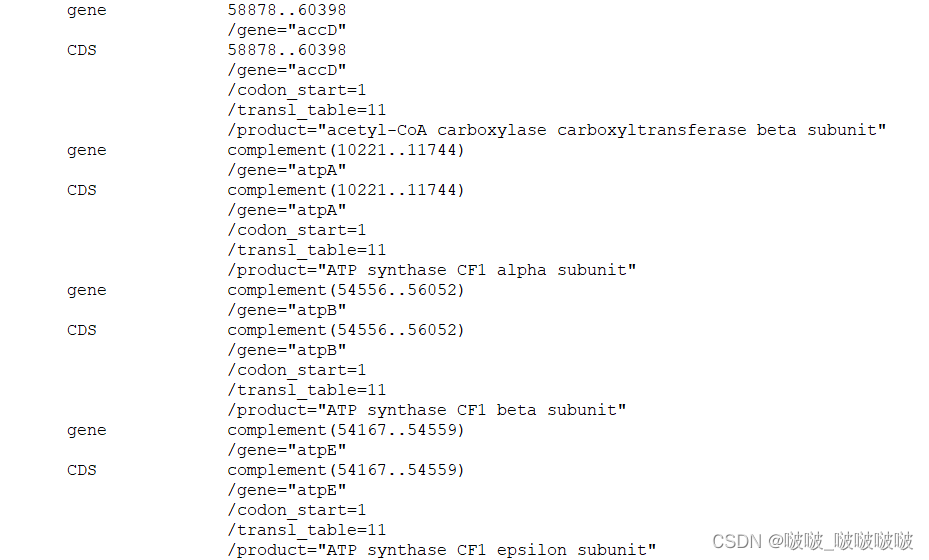
和NCBI文件相比,PGA注释的基因是乱序的 而且缺少了一个translation标签
使用biopython编写简单脚本可以实现该功能。
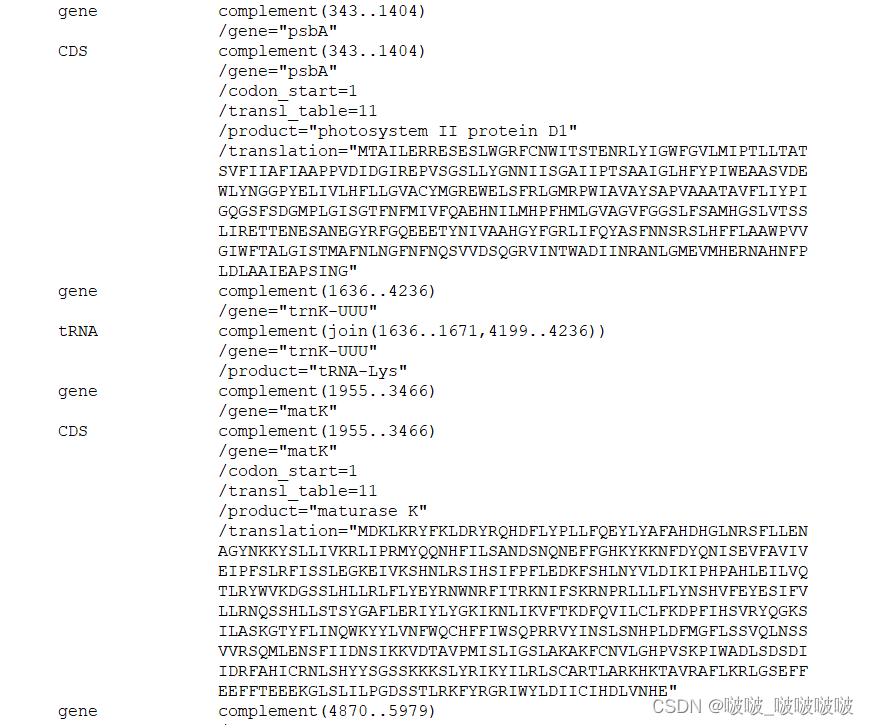
只需要写个小脚本进行排序然后添加数据translation数据标签即可
写在结尾
PGA注释的结果准确度还是挺高的,目前没有100%注释准确的软件 所有的结果都需要进行手动修改。
啵啵啵啵的学习笔记
公众号会经常分享一些叶绿体基因组相关的文章和脚本
最后
以上就是忧郁小松鼠最近收集整理的关于叶绿体基因组注释PGA篇叶绿体基因组PGA注释的全部内容,更多相关叶绿体基因组注释PGA篇叶绿体基因组PGA注释内容请搜索靠谱客的其他文章。
本图文内容来源于网友提供,作为学习参考使用,或来自网络收集整理,版权属于原作者所有。








发表评论 取消回复