前言
本文主要演示GEO数据库的一些工具,使用的数据是2015年在Nature Communications上发表的文章Regulation of autophagy and the ubiquitin-proteasome system by the FoxO transcriptional network during muscle atrophy.[pubmed:25858807]
作者通过将FoxO1-3-4-floxed小鼠(FoxO1,3,4 f / f)与表达Cre重组酶的转基因系在MLC1f启动子的控制下交叉,在肌肉中特异性地产生敲除的FoxO 1,3,4以产生肌肉特异性FoxO1,3,4三重敲除小鼠。这些小鼠要么自由进食,要么饥饿,随后分别提取4种情况小鼠的RNA,使用Affymetrix提供的试剂盒并根据标准Affymetrix方案制备,标记并与Affymetrix Mouse Genome 430 2.0 Arrays杂交cRNA,分析腓肠肌的基因表达。
GEO数据库筛选差异基因
首先,打开NCBI(https://www.ncbi.nlm.nih.gov/),如下图所示选择GEO Datasets,输入GDS5656,点击Search。
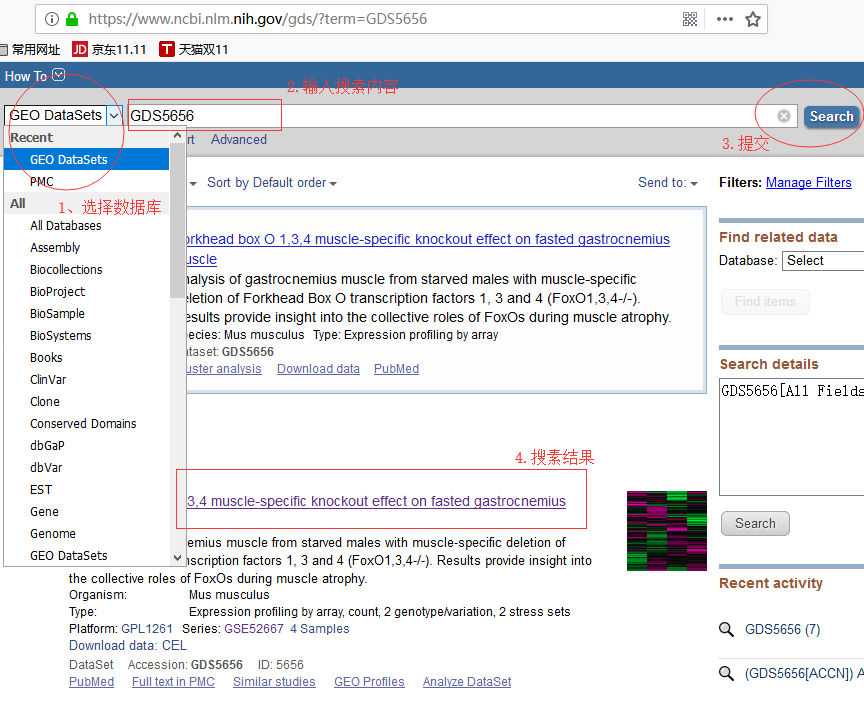
点击搜索到的结果
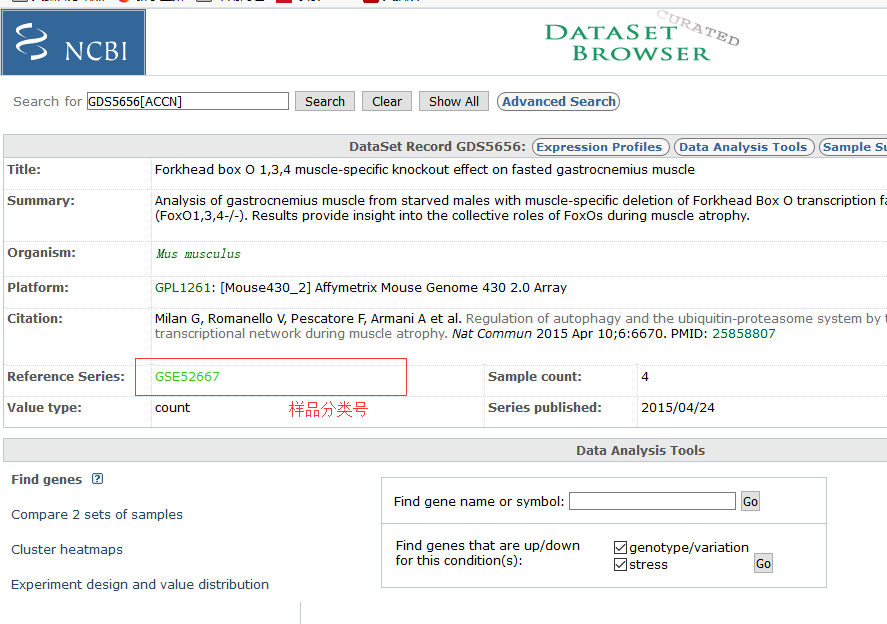
点击样品分类号,我们可以看到该研究的详情,包括文章研究内容、实验方案设计、样本详情等。

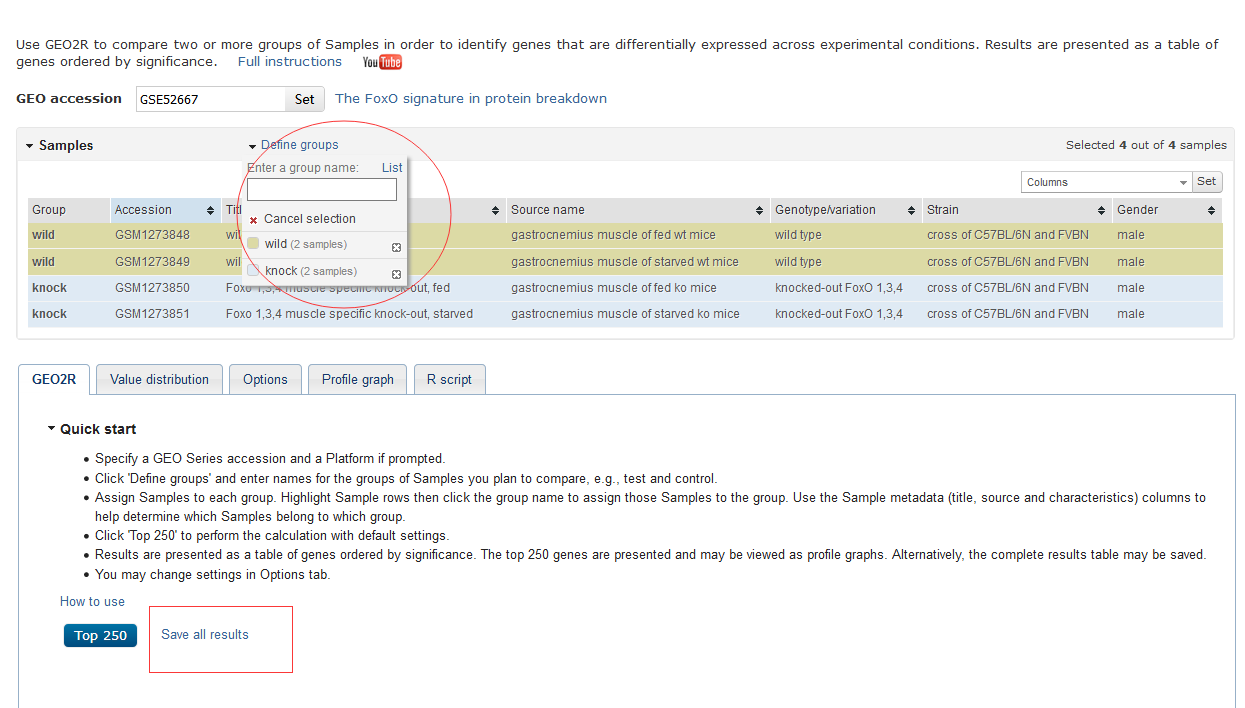
点击Analyze with GEO2R,利用在线工具进行数据分析。将4个样本分成了两组,分组完毕后,点击save all results,获取两组之间的差异表达基因。
得到如下所示的文本内容,将其粘贴到记事本(例如,保存为result.txt),然后导入到excel中(数据→自文本,选择result.txt文件导入),准备进行筛选。
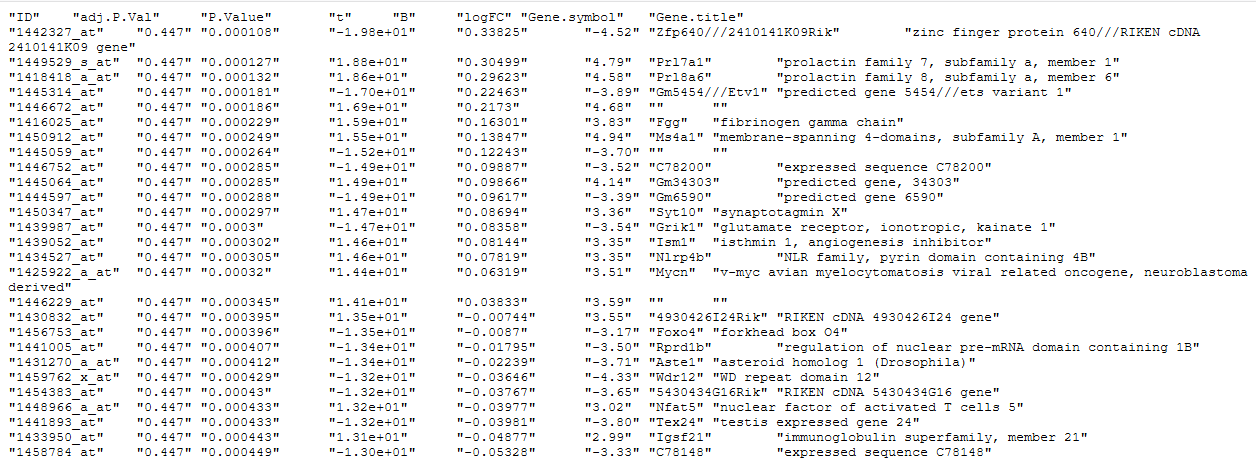
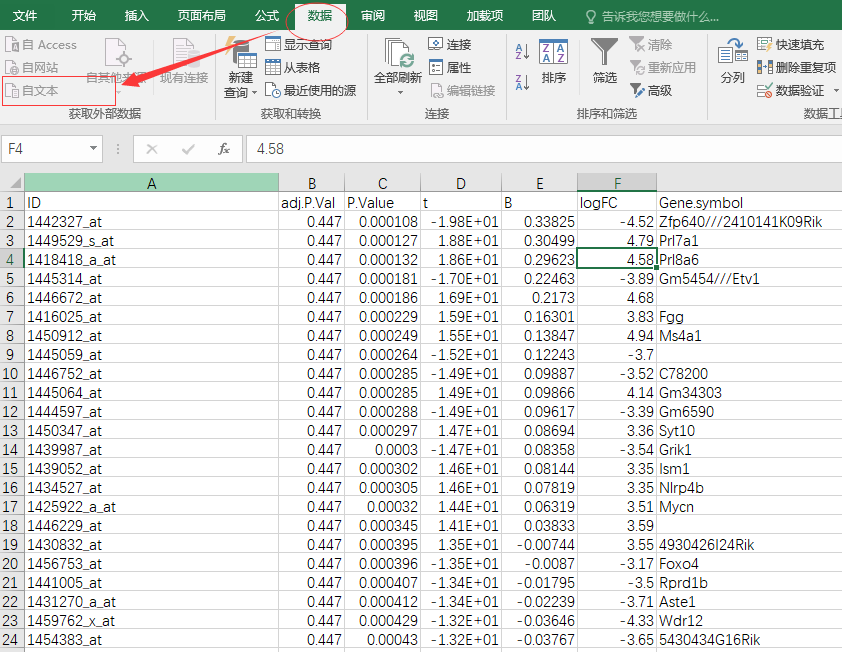
下一步,我们需要对差异表达基因的数据进行进一步的筛选。在这里,我们将p.value(p值,象征着差异的显著性)和logFC(log2处理过的fold change值,象征着差异的倍数)设定为: p.value<0.01, logFC<-2 or logFC>2。即差异表达非常显著,并且差异表达在4倍或-4倍以上(原文使用的是1.5倍阈值)。具体做法参见下图。
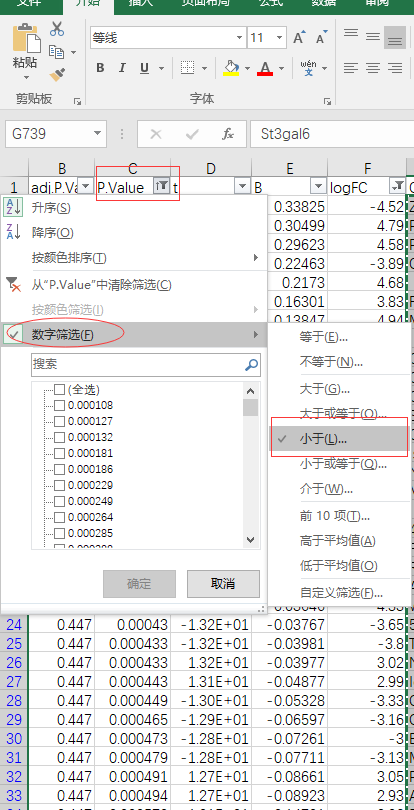
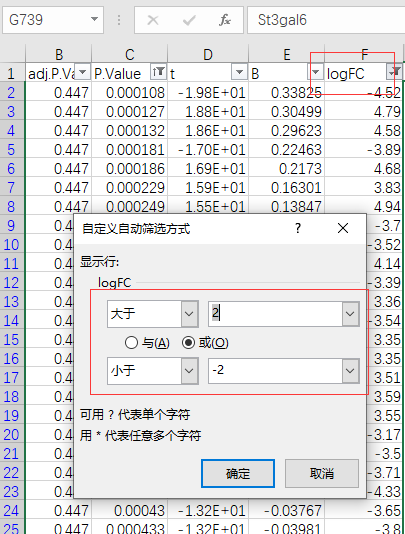
最后我们可以在EXCEL左下角的状态栏看到,一共筛选出来738个条目。
使用BioDBnet将geneSybol转换为Ensembl Gene ID
biodbnet(https://biodbnet-abcc.ncifcrf.gov/db/) db2db工具支持多个数据库gene id之间转换
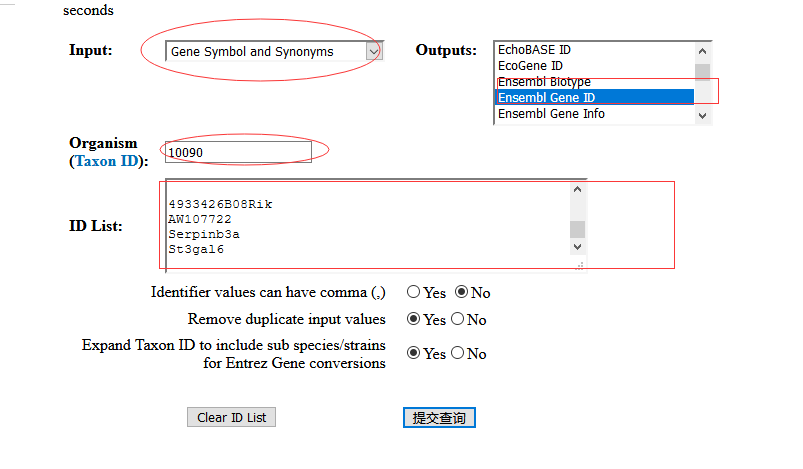
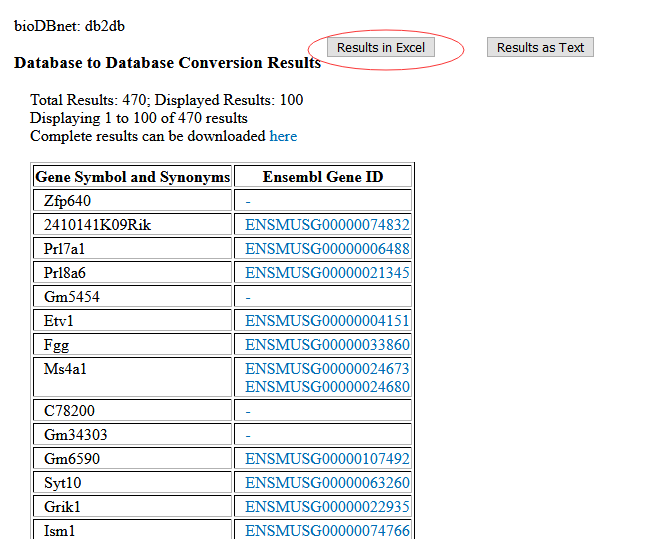
使用BioDBnet将geneSybol转换为Ensembl Gene ID,下载转换完的结果
使用KOBAS进行KEGG注释分析
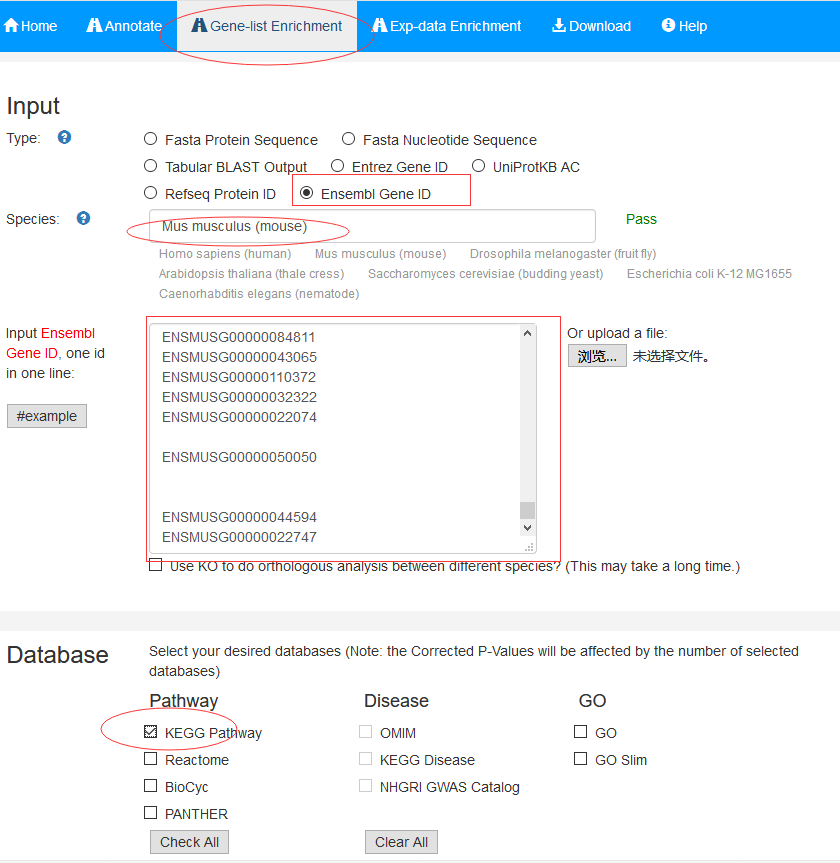
1. 输入类型选择:Ensembl Gene ID
2. 物种选择:Mus musculus(mouse)
3. 粘贴Ensembl Gene ID列表
4. 数据库 Clear All取消Pathway、Disease、GO全部选项,只选择KEGG Patway
点击RUN
分析结果链接:http://kobas.cbi.pku.edu.cn/result_kobas.php?taskid=181104291408457
下载
转载于:https://www.cnblogs.com/yahengwang/p/9906093.html
最后
以上就是壮观耳机最近收集整理的关于使用GEO数据库来筛选差异表达基因,KOBAS进行KEGG注释分析前言GEO数据库筛选差异基因使用BioDBnet将geneSybol转换为Ensembl Gene ID 使用KOBAS进行KEGG注释分析的全部内容,更多相关使用GEO数据库来筛选差异表达基因,KOBAS进行KEGG注释分析前言GEO数据库筛选差异基因使用BioDBnet将geneSybol转换为Ensembl内容请搜索靠谱客的其他文章。








发表评论 取消回复