分享一篇 Science 里不同批次的单细胞数据整合及批次校正方法
[1] Zheng L, Qin S, Si W, Wang A, Xing B, Gao R, Ren X, Wang L, Wu X, Zhang J, Wu N, Zhang N, Zheng H, Ouyang H, Chen K, Bu Z, Hu X, Ji J and Zhang Z 2021 Pan-cancer single-cell landscape of tumor-infiltrating T cells Science 374 abe6474

文章来自Science 上周刚发表的一批篇题为泛癌症T细胞单细胞图谱的文章。我们主要学习一下这篇文章的数据整合及批次校正方法。
处理流程图示
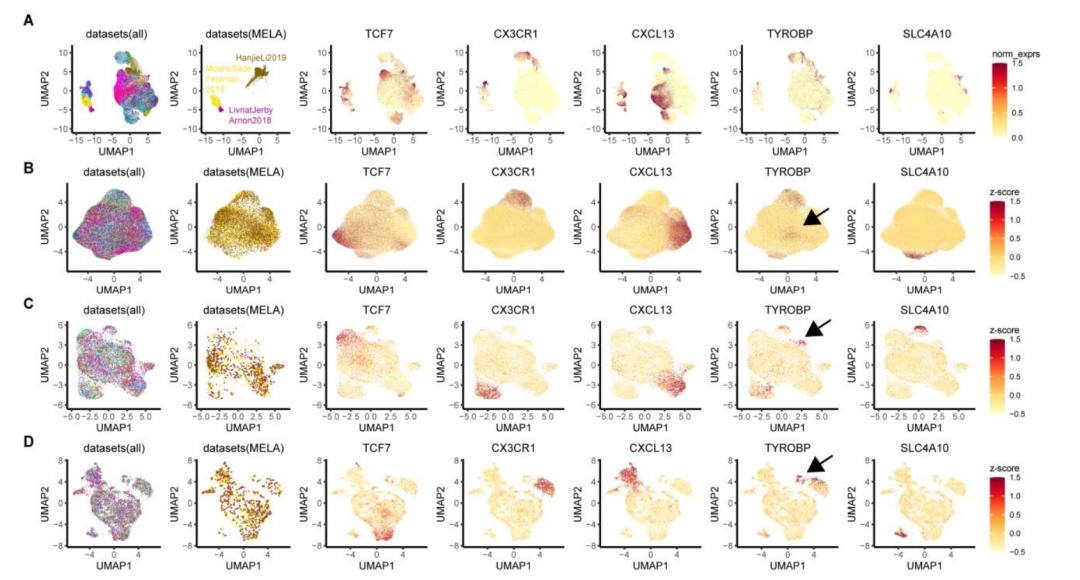
CD8+T细胞数据整合。
A.UMAP图显示CD8+ T细胞。每个点代表一个单细胞。第一张图显示的是来自所有数据集的细胞,第二张图显示的是黑色素瘤研究中的细胞。这两幅图都由数据集着色。很明显,来自不同数据集的细胞形成了不同的簇,说明批次效应的存在。下面的图显示了所有数据集的细胞,但由5个标记基因的标准化表达着色。
B.对A数据进行z-score标准化后的UMAP图。
C.对B数据应用mini-clusters处理后的UMAP图,也就是说,每个点代表一个小型集群。
D.对C数据应用批次矫正算法Harmony之后的UMAP图。
E.箱线图显示了A、B、C、D数据中的LISI (Local Inverse Simpson’s Index)分布。值得注意的是,在步骤B中,大部分细胞存在LISI > 1,提示未观察到明显的批量效应;在步骤B、C和D中,LISI显著增加,表明改进了数据整合。
数据整合和元集群识别
为了整合来自不同平台和不同研究的异构数据,采用了三个步骤。
对每个细胞进行
size-factor normalization,对每个基因进行z-score scaling为了减少技术噪音,如转录本退出,将单细胞划分为小组(称为微聚类,miniclusters),每个小组包含类似的细胞。这样,原始基因通过细胞表达矩阵转化为微聚类表达矩阵的基因。将所有数据集的矩阵按列组合,只保留所有数据集中存在的基因。合并的矩阵将用于下游分析。
应用批量效应校正算法Harmony对批次效应进行校正。
使用局部逆辛普森指数(local inverse Simpson’s Index,LISI)来评估整合效果。LISI定义了单个细胞附近的数据集的有效数量。LISI值越高,说明邻域的数据集越多,批处理效应越小。从之前的图可以看出z-score scaling 已经消除了由于平台和研究的差异而引起的批量效应,UMAP上数据集的均匀分布和LISI分布表明大多数细胞的LISI大于1证明了这一点。将单个细胞分组成小簇后,某些隐藏的细胞状态变得明显,并显著提高了LISI。使用Harmony进一步显著增加了LISI。
具体实现方法参阅原文附录。
往期
跟着Nature学作图 | 配对哑铃图+分组拟合曲线+分类变量热图
(免费教程+代码领取)|跟着Cell学作图系列合集
跟着Nat Commun学作图 | 1.批量箱线图+散点+差异分析
跟着Nat Commun学作图 | 2.时间线图
跟着Nat Commun学作图 | 3.物种丰度堆积柱状图
跟着Nat Commun学作图 | 4.配对箱线图+差异分析
最后
以上就是俊逸水蜜桃最近收集整理的关于分享一篇 Science 里不同批次的单细胞数据整合及批次校正方法分享一篇 Science 里不同批次的单细胞数据整合及批次校正方法的全部内容,更多相关分享一篇内容请搜索靠谱客的其他文章。








发表评论 取消回复