本文首发于我的个人博客, http://xuzhougeng.top/ 往期回顾: 在ArchR中,类似于UMAP和t-SNE的嵌入方法被用于在降维空间中可视化展示单细胞数据。这些嵌入有各自的优势和缺陷。我们之所以称它们为"嵌入"是因为他们只限于对聚类进行可视化而非用于鉴定聚类(在LSI子空间中的聚类分析)。UMAP和t-SNE的主要区别在于对细胞和聚类间的距离解释,t-SNE用于保留数据的局部结构,而UMAP则是保留局部结构的同时尽可能保留全局结构。从理论上来讲,UMAP中细胞聚类间的距离存在意义,而t-SNE则没有。也就是说,你不能说t-SNE中聚类A比聚类C更接近聚类B,而UMAP在设计的时候允许这种类型的比较。不过需要注意的是,由于UMAP是新出现的方法,因此使用UMAP的文章才刚刚兴起。 需要注意的是,无论是UMAP还是t-SNE,两个的运行结果都不是确定性的,也就是相同输入会得到不完全相同的结果。尽管如此,我们使用样本重复后发现t-SNE比UMAP更加的随机。此外,使用 无论您选择哪种方法,输入参数都会对结果嵌入产生剧烈影响。因此,了解各种输入参数并调整这些参数以最好地满足您自己的数据需要是很重要的。ArchR实现了一组默认的输入参数,这些参数适用于大多数情况,但实际上没有一组参数可以直接套用,我们要根据不同的细胞数、复杂度和质量进行调整。 我们使用 使用 我们使用 除了根据"Sample"外,我们还可以根据上一张鉴定的"Cluster"进行上色 使用 用 我们还可以使用 同样用 t-SNE图可以用 和之前UMAP一样,我们同样使用 用 和之前UMAP的操作类似,我们可以将 用 在第4章中,我们通过 保持和之前UMAP嵌入一样的参数,只修改 用 相同的方法用t-SNE进行可视化 用
第6章: 单细胞嵌入
uwot包里UMAP时,设置相同的随机数种子会得到相同的结果。选择使用UMAP还是t-SNE是有细微差别的,但在我们手中,UMAP非常适合各种应用,这是我们对scATAC seq数据的标准选择。UMAP的性能也比t-SNE快。也许最重要的是,使用UMAP可以创建一个嵌入并将新样本投影到该嵌入中,而这在t-SNE中是不可能的,因为数据的拟合和预测是同时发生的。6.1 Uniform Manifold Approximation and Projection (UMAP)
addUMAP函数运行UMAPprojHeme2 <- addUMAP(
ArchRProj = projHeme2,
reducedDims = "IterativeLSI",
name = "UMAP",
nNeighbors = 30,
minDist = 0.5,
metric = "cosine"
)@操作符可以从ArchRProject中列出有哪些可用的embedding,如projHeme2@embeddingsplotEmbedding函数绘制UMAP图,设置embedding="UMAP"。通过修改colorBy和name来告诉ArchR使用给定哪个元信息矩阵的列对细胞进行上色。p1是按照样本进行上色p1 <- plotEmbedding(ArchRProj = projHeme2, colorBy = "cellColData", name = "Sample", embedding = "UMAP")p2 <- plotEmbedding(ArchRProj = projHeme2, colorBy = "cellColData", name = "Clusters", embedding = "UMAP")ggAlignPlots将这两个图共同展示,"type=h"表示两个图是水平排列ggAlignPlots(p1, p2, type = "h")
plotPDF()可以将保存图片的矢量版。plotPDF(p1,p2, name = "Plot-UMAP-Sample-Clusters.pdf", ArchRProj = projHeme2, addDOC = FALSE, width = 5, height = 5)plotEmbedding()可视化之前用scran聚类的结果p1 <- plotEmbedding(ArchRProj = projHeme2, colorBy = "cellColData", name = "Sample", embedding = "UMAP")
p2 <- plotEmbedding(ArchRProj = projHeme2, colorBy = "cellColData", name = "ScranClusters", embedding = "UMAP")
ggAlignPlots(p1, p2, type = "h")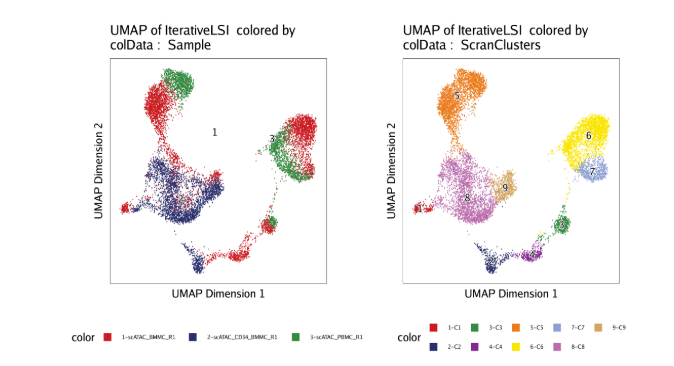
plotPDF()可以将保存图片的矢量版。plotPDF(p1,p2, name = "Plot-UMAP-Sample-ScranClusters.pdf", ArchRProj = projHeme2, addDOC = FALSE, width = 5, height = 5)6.2 t-Stocastic Neighbor Embedding (t-SNE)
addTSNE()函数运行得到projHeme2 <- addTSNE(
ArchRProj = projHeme2,
reducedDims = "IterativeLSI",
name = "TSNE",
perplexity = 30
)plotEmbedding()绘制t-SNE嵌入图。这里不需要考虑嵌入的类型,可以继续使用之前的colorBy和name参数p1 <- plotEmbedding(ArchRProj = projHeme2, colorBy = "cellColData", name = "Sample", embedding = "TSNE")
p2 <- plotEmbedding(ArchRProj = projHeme2, colorBy = "cellColData", name = "Clusters", embedding = "TSNE")
ggAlignPlots(p1, p2, type = "h")
plotPDF()可以将保存图片的矢量版。plotPDF(p1,p2, name = "Plot-TSNE-Sample-Clusters.pdf", ArchRProj = projHeme2, addDOC = FALSE, width = 5, height = 5)scran的结果和Seurat::FindClusters()的结果进行比较p1 <- plotEmbedding(ArchRProj = projHeme2, colorBy = "cellColData", name = "Sample", embedding = "TSNE")
p2 <- plotEmbedding(ArchRProj = projHeme2, colorBy = "cellColData", name = "ScranClusters", embedding = "TSNE")
ggAlignPlots(p1, p2, type = "h")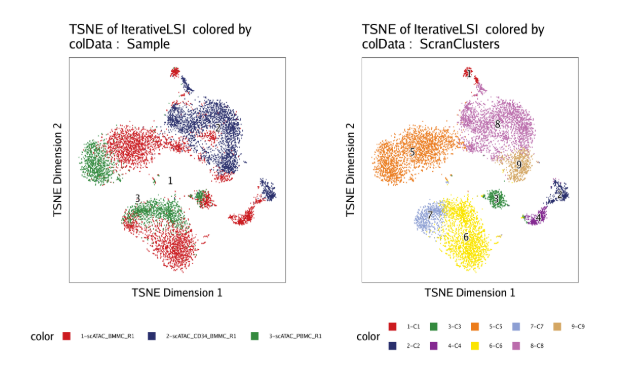
plotPDF()可以将保存图片的矢量版。plotPDF(p1,p2, name = "Plot-tSNE-Sample-ScranClusters.pdf", ArchRProj = projHeme2, addDOC = FALSE, width = 5, height = 5)6.3 Harmony后降维
addHarmony调用Harmony对数据进行批次校正,创建了一个名为"Harmony"的reducedDims对象。我们通过UMAP或t-SNE对结果进行嵌入可视化,对迭代LSI结果和Harmony校正结果进行比较,评估Harmony的作用。reducedDims="Harmony"projHeme2 <- addUMAP(
ArchRProj = projHeme2,
reducedDims = "Harmony",
name = "UMAPHarmony",
nNeighbors = 30,
minDist = 0.5,
metric = "cosine"
)
p3 <- plotEmbedding(ArchRProj = projHeme2, colorBy = "cellColData", name = "Sample", embedding = "UMAPHarmony")
p4 <- plotEmbedding(ArchRProj = projHeme2, colorBy = "cellColData", name = "Clusters", embedding = "UMAPHarmony")
ggAlignPlots(p3, p4, type = "h")
plotPDF()可以将保存图片的矢量版。plotPDF(p1,p2,p3,p4, name = "Plot-UMAP2Harmony-Sample-Clusters.pdf", ArchRProj = projHeme2, addDOC = FALSE, width = 5, height = 5)projHeme2 <- addTSNE(
ArchRProj = projHeme2,
reducedDims = "Harmony",
name = "TSNEHarmony",
perplexity = 30
)
p3 <- plotEmbedding(ArchRProj = projHeme2, colorBy = "cellColData", name = "Sample", embedding = "TSNEHarmony")
p4 <- plotEmbedding(ArchRProj = projHeme2, colorBy = "cellColData", name = "Clusters", embedding = "TSNEHarmony")
ggAlignPlots(p3, p4, type = "h")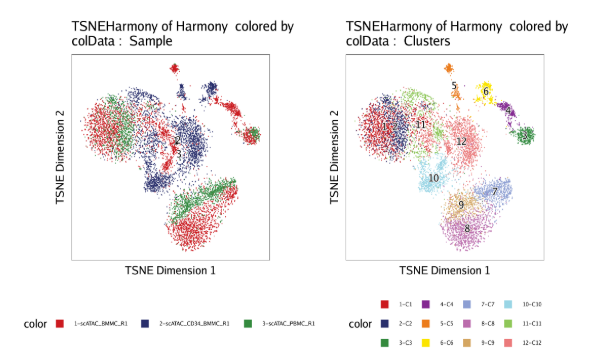
plotPDF()可以将保存图片的矢量版。plotPDF(p1,p2,p3,p4, name = "Plot-TSNE2Harmony-Sample-Clusters.pdf", ArchRProj = projHeme2, addDOC = FALSE, width = 5, height = 5)
最后
以上就是悦耳画笔最近收集整理的关于使用ArchR分析单细胞ATAC-seq数据(第六章)第6章: 单细胞嵌入的全部内容,更多相关使用ArchR分析单细胞ATAC-seq数据(第六章)第6章:内容请搜索靠谱客的其他文章。








发表评论 取消回复