生长曲线拟合–公式详细推导与matlab程序
写作目的
网上缺少详细的分析基于测量OD拟合Logistic方程的资料。
公式中具体的生物学意义与详细推导过程缺少汇总的文章。
经常遗忘公式的来源,对过程不熟悉,本文主要强化个人知识分享整理的内容。
其他生物学模型,可以过往发的博客 例如:https://blog.csdn.net/qq_33980829/article/details/110082048
本文主要内容
- 基于测量的 O D 600 OD_{600} OD600进行生长曲线的拟合
- 详细的公式推导过程
- 完整的matlab代码以及详细的代码注释
- 对公式中涉及的生物学意义进行说明
持续更新
- 对不同条件下生长曲线进行聚类分析
公式推导说明
- 公式基于公开的逻辑回归方程,本文主要是细化具体的推导过程
- 推导过程主要是给自己做个记录(个人需求)
- 如果对推导过程有问题,可以留言说明。
- 推导过程与代码实际运行有出入
公式推导
Logistic模型的微分表达式
1
N
i
∂
N
i
∂
t
=
r
(
1
−
N
i
K
)
(
1
)
frac{1}{N_{i}}frac{partial N_{i}}{partial t}=r({1-tfrac{N_{i}}{K}}) qquad (1)
Ni1∂t∂Ni=r(1−KNi)(1)
将右边移项到左边构成
( 1 − N i K ) ∗ 1 N i ∂ N i = r ∂ t ( 2 ) ({1-frac{N_{i}}{K}})*frac{1}{N_{i}}{partial N_{i}}=rpartial t qquad (2) (1−KNi)∗Ni1∂Ni=r∂t(2)
两边同时积分
− ( 1 K − N i − 1 N i ) ∂ N i = ∫ r ∂ t ( 3 ) -(frac{1}{K-N_{i}}-frac{1}{N_{i}}){partial N_{i}}=int r{partial t } qquad (3) −(K−Ni1−Ni1)∂Ni=∫r∂t(3)
l n K − N i N i = − r t + c ( 4 ) ln frac{K-N_{i}}{N_{i}}=-rt+c qquad (4) lnNiK−Ni=−rt+c(4)
两边同时取e的对数
K − N i N i = c ∗ e − r t ( 5 ) frac{K-N_{i}}{N_{i}}=c*e^{-rt}qquad (5) NiK−Ni=c∗e−rt(5)
K N i = c ∗ e − r t + 1 ( 6 ) frac{K}{N_{i}}=c*e^{-rt}+1qquad (6) NiK=c∗e−rt+1(6)
两边取倒数,再两边乘K
N i = K c ∗ e − r t + 1 ( 7 ) {N_{i}}= frac{K}{c*e^{-rt}+1}qquad (7) Ni=c∗e−rt+1K(7)
参数说明
- r r r 不同资源下的最大生长速率 K K K 在特定资源下的特征生长速率。c为微分常数
- 有文献说 r与k的大小差异能反映进化过程中策略选择。关于r策略与k策略详细见博客{1}中的文章
matlab代码程序
%% 生长曲线拟合
clc
clear
close all
[~, ~, raw] = xlsread('C:UsershuangDesktop生长.xlsx','Sheet1','C2:M16'); %导入文件
%% 创建输出变量
U_1= reshape([raw{:}],size(raw));
%% 清除临时变量
clearvars raw;
U_1(:,2:end)=U_1(:,2:end)/1000;
a_num=1;%标记数值
for i=4:size(U_1,2) %输入要拟合数据内容
x=U_1(:,1);
y=U_1(:,i);
fx=@(b,x)(b(1)./(1+b(2).*exp(-b(3).*x)));
%x=xlsread('data','a1:b111');输入第一组数据
% data1
% plot(x,y,'o','markerfacecolor','r')
b=[1 0.6 4.3]; %初始迭代值 最大值 生长速率 (根据具体实验来设定,初始值在本方程拟合影响不大)
for l=1:30 %拟合过程迭代
b=lsqcurvefit(fx,b,x,y);
b=nlinfit(x,y,fx,b);
end
n=length(y);
disp('data1')
SSy=var(y)*(n-1);
y1=fx(b,x);
RSS=(y-y1)'*(y-y1);
Rsquare(1,a_num)=(SSy-RSS)/SSy;
x1=linspace(min(x),max(x),300);
y1=fx(b,x1);
plot(x,y,'*')
hold on
b_num(a_num,:)=b;%保存每次拟合的系数信息
a=strcat(num2str(b(1)),'./(1+',num2str(b(2)),'.*exp(-',num2str(b(3)),'.*x))'); %拟合的函数
fplot(a,[min(x),max(x)],'linewidth',1)% 绘制拟合的函数
% plot(x1,y1,'-','linewidth',1) %绘制曲线
% plot(x1,y1,'b-','linewidth',2.5)
hold on
% plot(x,y,'o')
% plot(x,y,'o','markerfacecolor','r')
% tex=texlabel(strcat(num2str(b(1)),'./(1+',num2str(b(2)),'.*exp(-',num2str(b(3)),'.*x))'));%公式表示
% text(5,1.1,tex) %文本位置
hold on
a_num=a_num+1;
end
xlabel('生长时间(h)','Interpreter','LaTex')
ylabel('OD$_{600}$','Interpreter','LaTex')
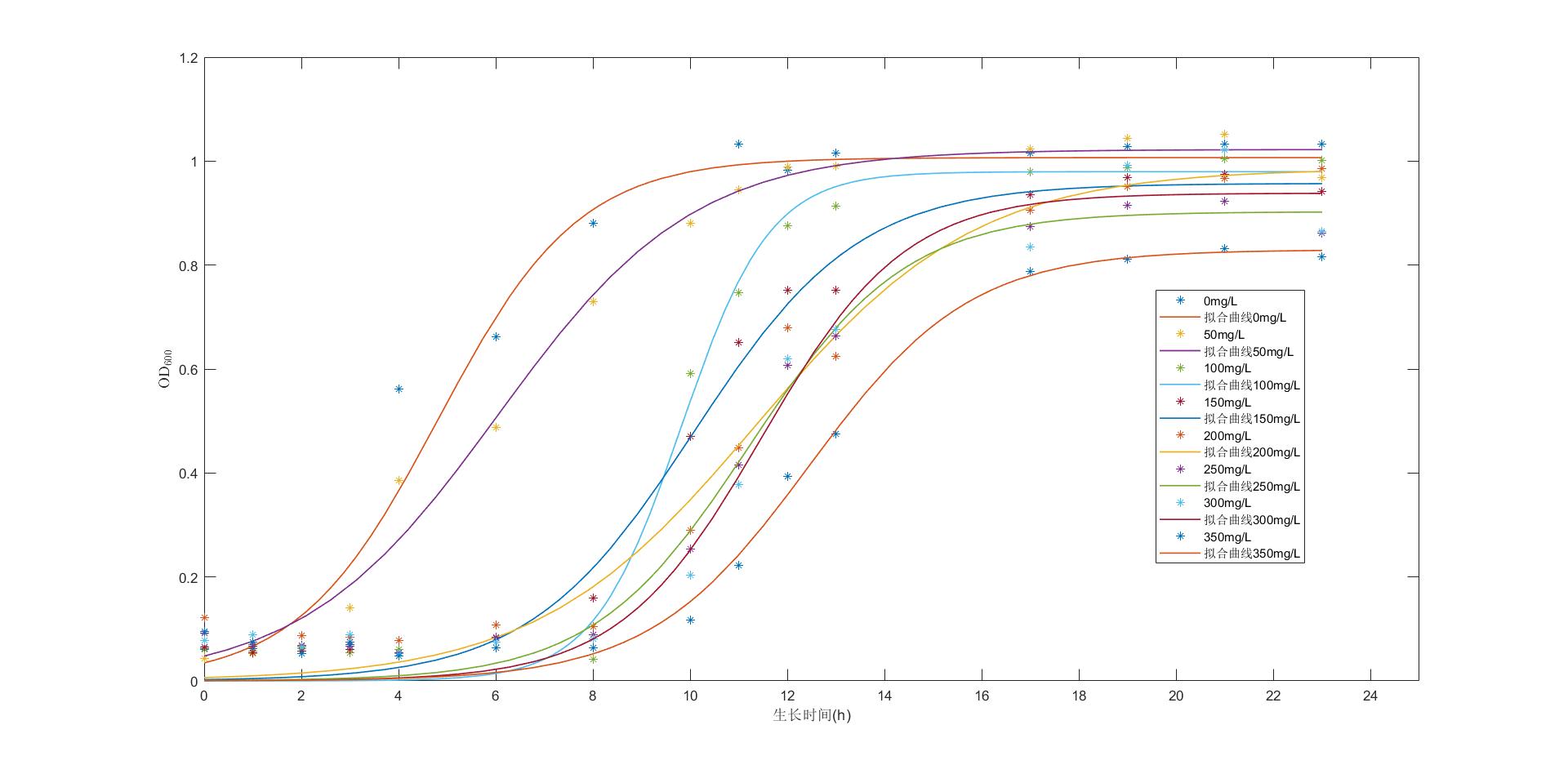
legend('0mg/L','拟合曲线0mg/L','50mg/L','拟合曲线50mg/L','100mg/L','拟合曲线100mg/L','150mg/L','拟合曲线150mg/L',...
'200mg/L','拟合曲线200mg/L','250mg/L','拟合曲线250mg/L','300mg/L','拟合曲线300mg/L','350mg/L','拟合曲线350mg/L')
% legend('Position',[0.75928385140126 0.341278249168987 0.0951822931443652 0.312833518330545])
xticks(min(x):2:max(x)+2); %x坐标区间范围以及间隔
hold off
%% 不同生长OD聚类
figure
% y_1=b_num(:,1);
% plot(b_num(:,1),b_num(:,3),'*')
% ylim([0.3,max(b_num(:,3))])
% yticks([0.3:0.1:max(b_num)]);
a=[1:8];
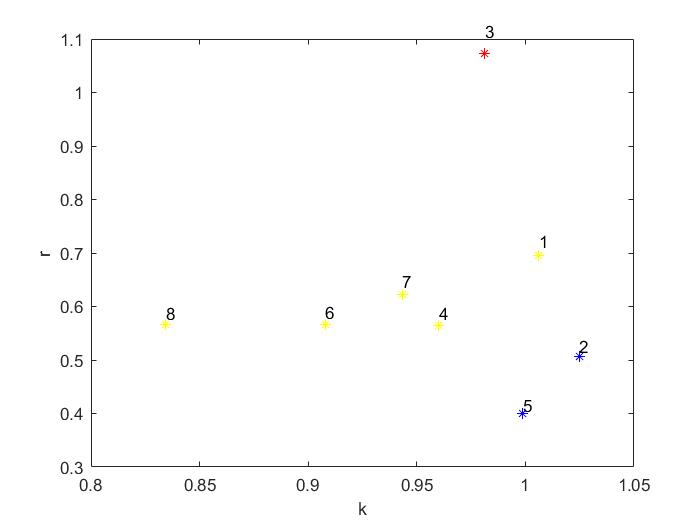
[cidx, ctrs] =kmeans(b_num(:,1:2:3),3);
plot(b_num(cidx==1,1),b_num(cidx==1,3),'r*', ...
b_num(cidx==2,1),b_num(cidx==2,3),'b*',b_num(cidx==3,1),b_num(cidx==3,3),'y*');
a=num2cell(a);
text(b_num(:,1),b_num(:,3)*1.04,a)
xlabel('k')
ylabel('r')
代码运行结果
生长曲线拟合
拟合的曲线聚类
数据展示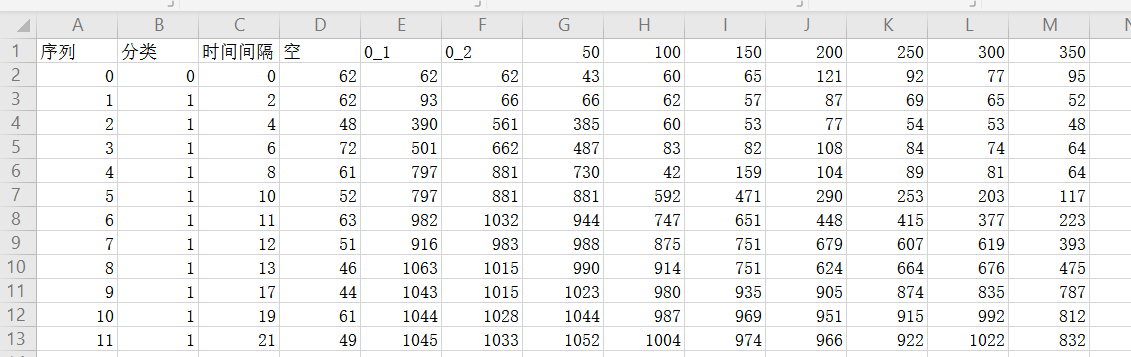
最后
以上就是高贵乌龟最近收集整理的关于微生物生长曲线拟合--公式详细推导与matlab程序生长曲线拟合–公式详细推导与matlab程序的全部内容,更多相关微生物生长曲线拟合--公式详细推导与matlab程序生长曲线拟合–公式详细推导与matlab程序内容请搜索靠谱客的其他文章。








发表评论 取消回复