智源导读:在生物医学领域,将深度学习用于蛋白结构预测成为近年大热门。在6月3日智源大会的AI赋能生命健康与生物医药论坛上,北京大学教授马剑竹做了题为“结构性和功能性基因组学的机器学习算法”的报告,阐述了将人工智能技术用于蛋白质结构和功能预测的相关工作。
整理:肖健
讲者简介:马剑竹,现担任北京大学人工智能研究院副教授,此前曾任美国普渡大学生物化学系、计算机系助理教授。马博士是机器学习领域的计算机专家,主要研究科学、医学及卫生领域中数据带来的挑战。马博士曾发表多篇生物医学应用相关论文(收录于《Nature Methods》《Nature Cancer》 《Cell》《Nature Communications》 《PNAS》等),其论文、发言、海报及研究曾在RECOMB、ISMB等顶级会议中获奖。
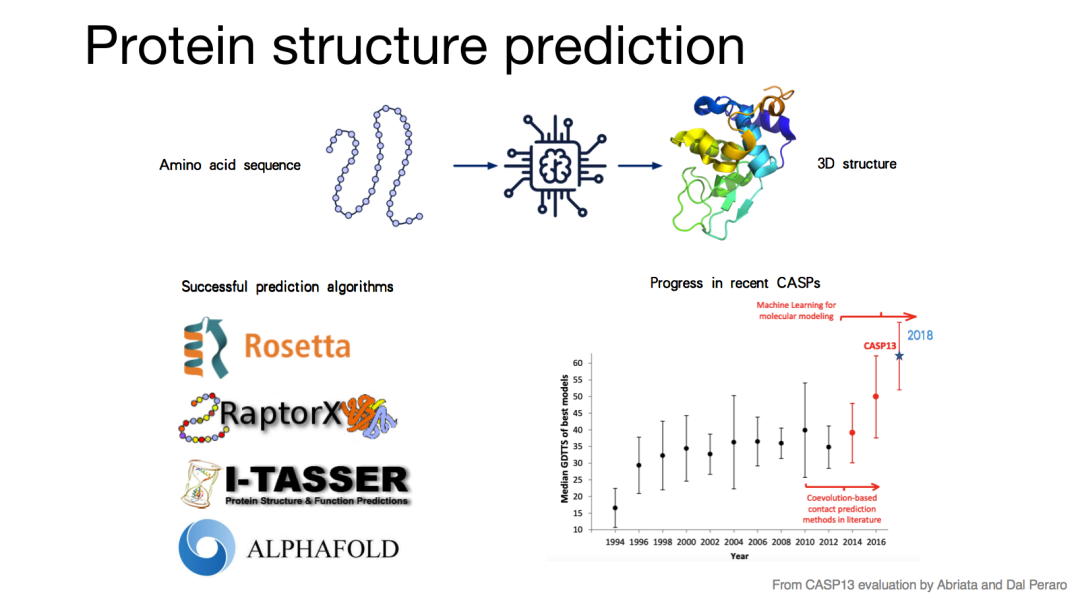
近年来,从氨基酸序列预测蛋白质空间结构成为热点的研究。基于同源蛋白模板匹配预测空间结构,是主流的预测方法。与进化过程蛋白质序列变化相比,蛋白质空间结构的变化更具有保守性,因此存在多种蛋白质序列对应相似的空间结构。因此,提出一种蛋白质序列空间距离表示对于蛋白质空间结构预测非常重要。
RaptorX是首个将深度学习用于蛋白结构预测软件,曾在CASP12和CASP13中接触预测排名第一。RaptorX软件由马教授与伊利诺伊大学厄巴纳-香槟分校计算机科学系副教授彭健共同开发。
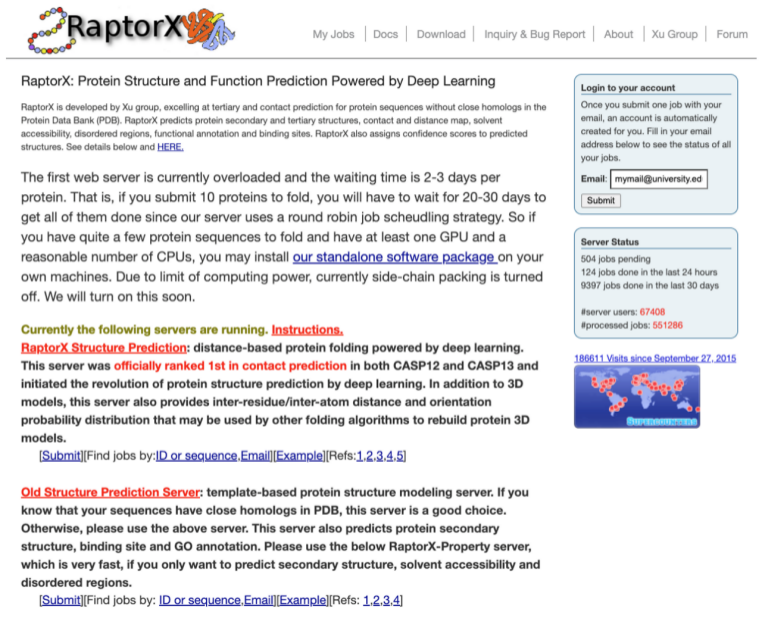
图(1)RaptorX软件的官方主页
目前已有的蛋白质数据库中有约17万的蛋白质结构,但实际只有约1000种蛋白结构类别,因此任意一种新的蛋白质,几乎都可以在数据库中找到与之类似结构的蛋白,所以可以用数据比对的方式,从数据库中找到一个或多个模板,进而在模板的基础上预测未知蛋白的结构。
然而,实际上序列比对是一件很难的事。有些蛋白质结构非常类似,但它们的序列是完全不一样的,所以简单地用字符串匹配的方式做比对是行不通的。这个现象背后蕴含着生物学原理:蛋白质的结构比序列更加保守,即序列变化时仍能维持基本的蛋白结构不变。如何将不同的蛋白序列联配(Protein Alignment),得到氨基酸序列的一一映射,这就是马教授和彭教授曾一同研究的问题。这一问题在今天仍有重要的意义,因为AlphaFold2中也使用的同源蛋白的信息,找同源蛋白的过程也使用了蛋白联配技术。
做两个蛋白质的联配可以用动态规划算法,填写动态规划表格,再回溯找到表格中的通路,也就是联配方式。但难点在于不知道某一个字母应当对应哪个字母,因此需要用监督学习为联配结果赋分。于是可以将已知结构的蛋白做堆叠,得到真实的联配方式,然后以最大似然作为优化目标,即让真实联配方式的分值最大化为目标来训练神经网络。这是一种基于格子的条件随机场方法(grid conditional random field),其中配分函数(Partition function)刚好能在多项式时间算出,因此可以对数似然函数做梯度下降。该论文“A conditional neural fields model for protein threading”发表在Bioinformatics上。
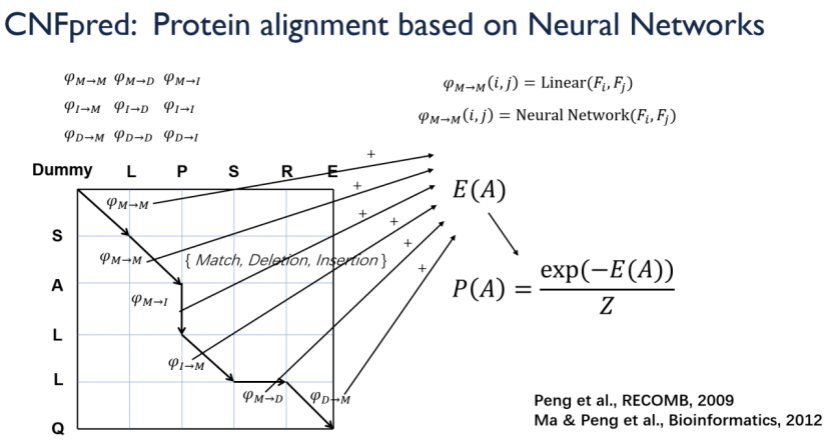
图(2)RaptorX背后基于深度学习预测蛋白质结构的算法
RaptorX背后的方法并没有触及“多种不同的蛋白质对应相似的空间结构”这一生物问题的固有性质,而是直接套用了机器学习算法。原有方法相当于是将现有的联配与真实的联配在每一节点作比较,相同则加分,不同则减分。但事实上,蛋白联配是两个几何体做堆叠,如果两个几何体错位得很少仍然可以认为它们结构相似,比如假设真实对应是1对1,2对2,3对3等,现有对应是1对2,2对3,3对4等,它们的结构看上去相差并不远;如果相差较远则联配得不好。
但原有方法不能捕捉这一信息,所以需要一种新的损失函数能够体现联配之间的几何差异。教授们想到一种精妙的方法,将现有的联配与真实的联配的距离用两种联配对应表格中路径围成的面积来度量,以面积最小为目标来训练神经网络。马剑竹教授的这项工作“PALM: Probabilistic Area Loss Minimization for Protein Sequence Alignment”发表在UAI上。
同时,马教授还抛出一个开放问题,如果要计算三个蛋白序列或多个的联配,马教授提供了一种初步的思路,仿照两序列联配问题,该情况下的联配需要以多维空间中的两条曲线围成的曲面的面积作为损失函数,同样以面积最小作为约束。
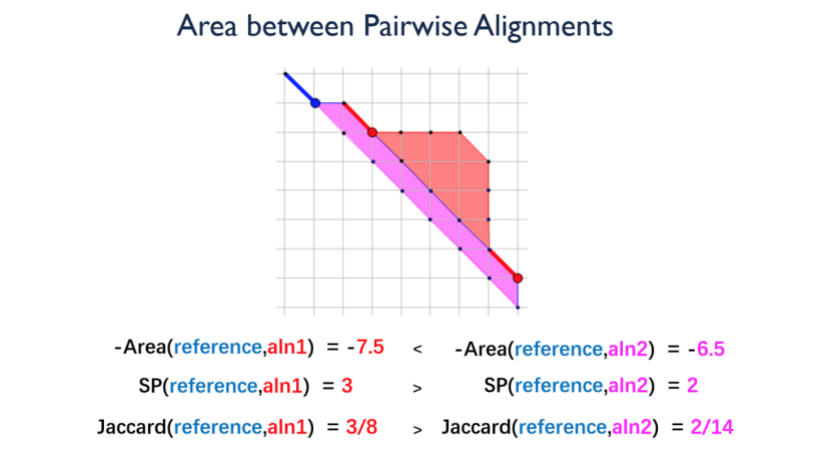
图(3)改进后的蛋白质结构预测算法
对于蛋白质功能预测问题,马教授提出了一种基于元学习的蛋白质功能预测方法。其中这种蛋白有很多族,每一族内的蛋白比较相似。现有的方法有两类,一种对每一族蛋白分别进行建模,一种把所有组都合在一起建模。前一种方法的问题是部分族的有标签数据很少,后一种方法会丢失族群间的区别。这在机器学习中是一种标准的多任务问题,这些任务间有关联性,又不太一样。利用元学习的思想,将每族的蛋白与短肽的结合作为一个任务,先对各任务做元学习模型,得到对不同族的蛋白非常敏感的表示,再将这一模型迁移到有标签数据很少的任务上。这项工作“Mitigating Data Scarcity in Protein Binding Prediction Using Meta-Learning”发表在RECOMB上。
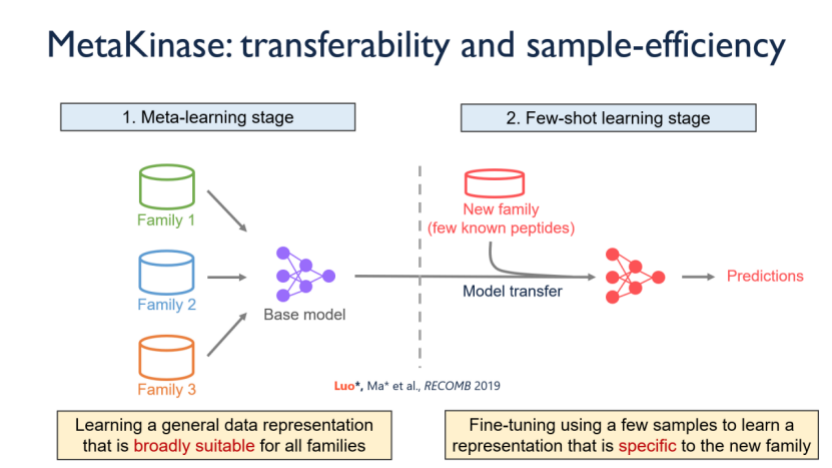
图(4)基于元学习的蛋白质功能预测模型
扫码加入「AI赋能生命健康与生物医药」交流群,参与相关话题讨论

最后
以上就是和谐时光最近收集整理的关于北大马剑竹 | 结构性和功能性基因组学的机器学习算法的全部内容,更多相关北大马剑竹内容请搜索靠谱客的其他文章。








发表评论 取消回复