
之前这篇文章(路人乙小明:用entrez eutilities来查pubmed文献)提过如何利用entrez eutilities查找文献。当时在最后面获取论文摘要和题录的部分直接用了text的模式。最近觉得用xml模式能获得更加细致的数据。比如我如果只想获得题目,杂志,一作,doi这几个信息,如果直接用text获取摘要,然后再用regex就比较容易出错,但是xml就不会有这样的问题(大概……)
况且用python重新写一下那段程序也是好的。
导入各种包
import requests
import os
import xml.etree.ElementTree as ET
import pyperclip
import argparse
import rerequests需要自己安装,是很方便的一个获取网络信息的工具。
os当时导进来主要是为了删除写入的临时文件(os.remove()),调试程序的时候总不能每次都从entrez读数据吧,一来比较慢,二来万一整天发请求会被封的。
xml.etree.ElementTree是python自带的xml解析工具
pyperclip是读取剪切板的工具
argparse是加入命令行参数的工具
re就是正则的包
参数和其他信息准备
reLink = re.compile(r'https://www.ncbi.nlm.nih.gov/pubmed/(d+)')
parser = argparse.ArgumentParser(
"-i pmid -l pubmed link")
parser.add_argument('-i')
parser.add_argument('-l')
parser.add_argument('-verbose')
parser.add_argument('-paste')
args = parser.parse_args()
if args.i is not None:
print(args.i)
ids = args.i
elif args.p is not None:
ids = re.search(reLink,pyperclip.paste()).group(1)
if ids is None:
raise Exception("link format is wrong")
elif args.l is not None:
print(args.l)
ids = re.search(reLink,args.l).group(1)
if ids is None:
raise Exception("link format is wrong")
else:
ids = '30693853'
dbname = 'pubmed'其实从entrez找文章的摘要,最核心的命令是
fetchedData = requests.get(
f"""https://eutils.ncbi.nlm.nih.gov/entrez/eutils/efetch.fcgi?db={dbname}&id={ids}&rettype=abstract&retmode=XML
"""
)需要提供的参数是下面4个:
- db:数据库名称,我们这里设置成pubmed
- id:记录条目的id,这里是指一个文章的pmid
- rettype:获取内容类型,这里使用abstract
- retmode:内容方式,这里使用XML
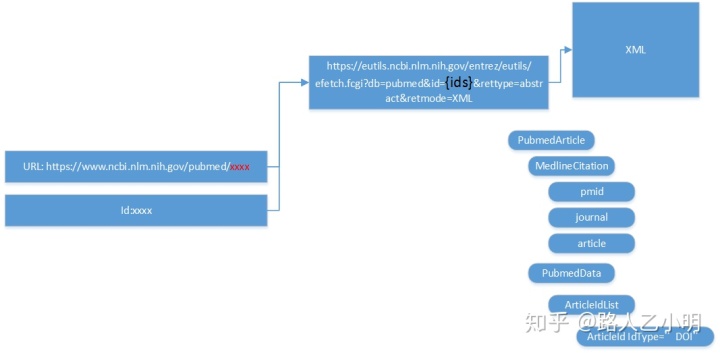
requests在get了entrez返回的数据以后:
with open('out.xml', 'w', encoding='utf8') as xmlFile:
xmlFile.write(fetchedData.text)
#调试完成以后直接读取requests get到的数据,out.xml只作暂存之用
rstDict = xmlParse(fetchedData.text)
#用format,按照输出格式将提取的数据合成文本
txt = """
title: {}
journal: {}, {}-{}, {}
author: {}, et al.
doi: {}
""".format(
rstDict['title'],
rstDict['journal']['title'],
rstDict['journal']['volume'],
rstDict['journal']['issue'],
rstDict['journal']['pubyear'],
rstDict['authors'][0],
rstDict['doi']
)
abstracts = "abstract:n".join(["{}:n{}".format(k, v)
for k, v in rstDict['abstract'].items()])
# 使用verbose参数决定是否输出摘要
if args.verbose is not None:
txt = txt+abstracts
pyperclip.copy(txt)
print(txt) xmlParse这个函数
整个程序主要的部分就是这个函数。我的实现方法比较naive,也没有对xml作安全性的防范。
def xmlParse(strtxt):
root = ET.fromstring(strtxt)
rstDict = {}
rstDict['pmid'] = root.find('./PubmedArticle/MedlineCitation/PMID').text
rstDict['journal'] = root.find('./PubmedArticle/MedlineCitation/Article/Journal')
rstDict['journal'] = {
'volume': rstDict['journal'].find('./JournalIssue/Volume').text,
'issue': rstDict['journal'].find('./JournalIssue/Issue').text,
'pubyear': rstDict['journal'].find('./JournalIssue/PubDate').text,
'title': rstDict['journal'].find('./Title').text,
'abbrev': rstDict['journal'].find('./ISOAbbreviation').text,
}
rstDict['title'] = root.find('./PubmedArticle/MedlineCitation/Article/ArticleTitle').text
rstDict['doi'] = [e for e in root.findall(
'./PubmedArticle/PubmedData/ArticleIdList/ArticleId') if e.get('IdType')=='doi'][0].text
rstDict['abstract'] = root.find(
'./PubmedArticle/MedlineCitation/Article/Abstract')
abstractLabels = [e.get('Label') for e in rstDict['abstract'].findall('AbstractText')]
abstractText = [
e.text for e in rstDict['abstract'].findall('AbstractText')]
rstDict['abstract'] = dict(zip(abstractLabels,abstractText))
rstDict['authors'] = root.find(
'./PubmedArticle/MedlineCitation/Article/AuthorList')
authorsList = rstDict['authors'].findall('Author')
rstDict['authors'] = ["{}, {}({})".format(
e.find('LastName').text,
e.find('ForeName').text,
e.find('Initials').text) for e in authorsList]
return rstDict这个函数主要由两类操作,一类就是直接能够通过text属性获取数据的,比如像rstDict['pmid']= root.find('./PubmedArticle/MedlineCitation/PMID').text这样的东西。这类只需要确定好xml节点的xpath,弄进find函数就没问题了。
另外一类,比如像authorsList这种,数量不定,返回的数据(rstDict['authors']) 是数据形式,就需要把find函数和findall函数联合起来用:
......
rstDict['authors'] = root.find(
'./PubmedArticle/MedlineCitation/Article/AuthorList')
authorsList = rstDict['authors'].findall('Author')
rstDict['authors'] = ["{}, {}({})".format(
e.find('LastName').text,
e.find('ForeName').text,
e.find('Initials').text) for e in authorsList]使用的时候可以先复制一个文章的url,,然后用的时候可以先复制一个文章的url,比如https://www.ncbi.nlm.nih.gov/pubmed/7450738/,然后 运行程序,外加一个-p的参数, 程序就可以读取剪贴板然后将简化的题录放回剪贴板。
代码全文在:https://gist.github.com/beneon/cd2bb92721c2c2c3fdb95f82d1df3b29
最后
以上就是矮小猫咪最近收集整理的关于如何用python自动获取文献_[python]eutilities获取文献题录的全部内容,更多相关如何用python自动获取文献_[python]eutilities获取文献题录内容请搜索靠谱客的其他文章。




![如何用python自动获取文献_[python]eutilities获取文献题录](https://www.shuijiaxian.com/files_image/reation/bcimg8.png)



发表评论 取消回复