因为R的rmda包做不了COX回归临床决策曲线,很多朋友都是通过ggdca包来绘制COX回归临床决策曲线,最近很多粉丝使用ggdca包来绘制COX回归临床决策曲线出现问题过来问我,我绘制的时候没发现什么问题,所以也回答不了,但是我看了一些别的公众号博主说是因为ggdca和survival包冲突,不能从R下载ggdca包,要从作者主页下载才可以,大家可以试一下。
好了,废话不多说,今天介绍R的dcurves包,这个包功能很全面,可以绘制logistic回归、COX回归、竞争风险模型等临床决策曲线,今天我们主要介绍COX回归临床决线,让大家多一种选择。继续使用我们的乳腺癌数据(公众号回复:乳腺癌可以获得该数据),dcurves包不能通过RStudio安装,会出错,我们直接通过R语言安装
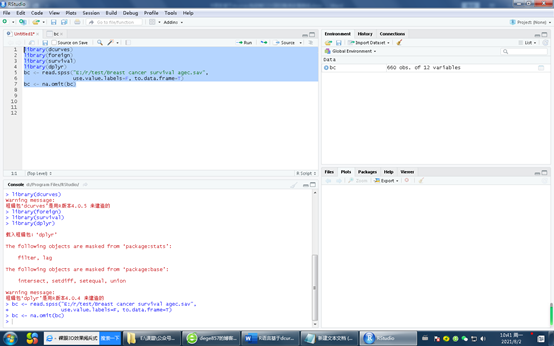
安装好以后就可以开始操作了,我们先导入R包和数据
library(dcurves)
library(foreign)
library(survival)
library(dplyr)
bc <- read.spss("E:/r/test/Breast cancer survival agec.sav",
use.value.labels=F, to.data.frame=T)
bc <- na.omit(bc)
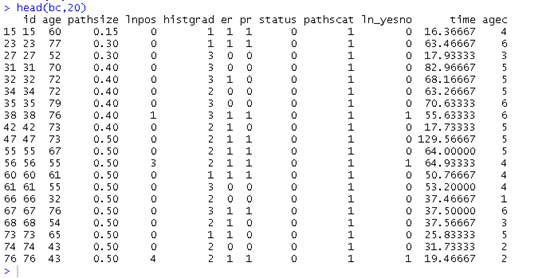
age表示年龄,pathsize表示病理肿瘤大小(厘米),lnpos表示腋窝淋巴结阳性,histgrad表示病理组织学等级,er表示雌激素受体状态,pr表示孕激素受体状态,status结局事件是否死亡,pathscat表示病理肿瘤大小类别(分组变量),ln_yesno表示是否有淋巴结肿大,time是生存时间,后面的agec是我们自己设定的,不用管它。
有部分变量为分类变量,我们先把它转换成因子
bc$histgrad<-as.factor(bc$histgrad)
bc$er<-as.factor(bc$er)
bc$pr<-as.factor(bc$pr)
bc$ln_yesno<-as.factor(bc$ln_yesno)
下面建立COX回归方程,假设我们想了解3年(36个月)生存的情况,我们建立3个COX回归模型(乱建的)
f1<-coxph(Surv(time,status)~er+histgrad+pr+age+ln_yesno,bc)
f2<-coxph(Surv(time,status)~er+histgrad+ln_yesno,bc)
f3<-coxph(Surv(time,status)~ln_yesno,bc)
建好以后,我们需算出每个模型的3年生存率
bc$pr_failuref136 = c(1- (summary(survfit(f1, newdata=bc), times=36)$surv))
bc$pr_failuref236 = c(1- (summary(survfit(f2, newdata=bc), times=36)$surv))
bc$pr_failuref336 = c(1- (summary(survfit(f3, newdata=bc), times=36)$surv))
我们可以分个画出每个模型的生存曲线
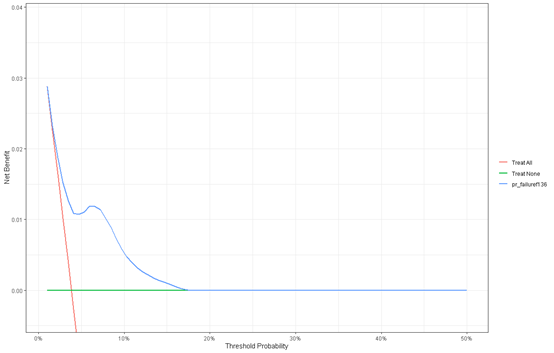
f1的
dca(Surv(time,status) ~ pr_failuref136,
data = bc,
time = 36,
thresholds = 1:50 / 100) %>%
plot(smooth = T)
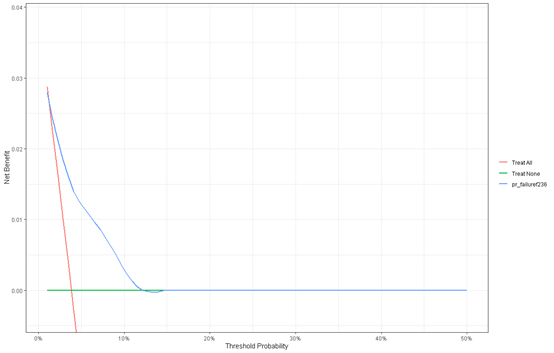
其他两个
dca(Surv(time,status) ~pr_failuref236,
data = bc,
time = 36,
thresholds = 1:50 / 100) %>%
plot(smooth = T)
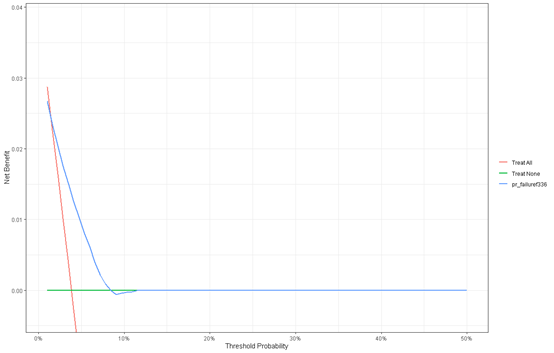
dca(Surv(time,status) ~pr_failuref336,
data = bc,
time = 36,
thresholds = 1:50 / 100) %>%
plot(smooth = T)
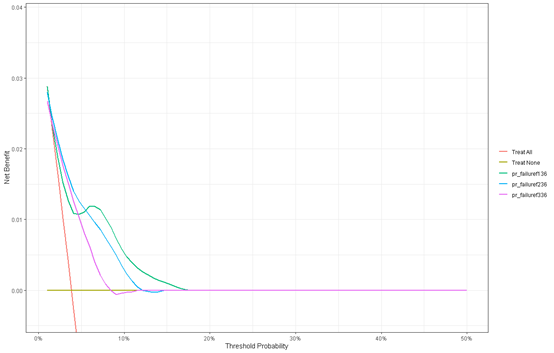
也可以画在一张图
dca(Surv(time,status) ~ pr_failuref136+pr_failuref236+pr_failuref336,
data = bc,
time = 36,
thresholds = 1:50 / 100) %>%
plot(smooth = T)
同理,我们想知道其他时间的生存情况也是一样,改改参数就可以了
5年生存率的决策曲线
bc$pr_failure60 = c(1- (summary(survfit(f1, newdata=bc), times=60)$surv))
dca(Surv(time,status) ~ pr_failure60,
data = bc,
time = 60,
thresholds = 1:50 / 100) %>%
plot(smooth = T)
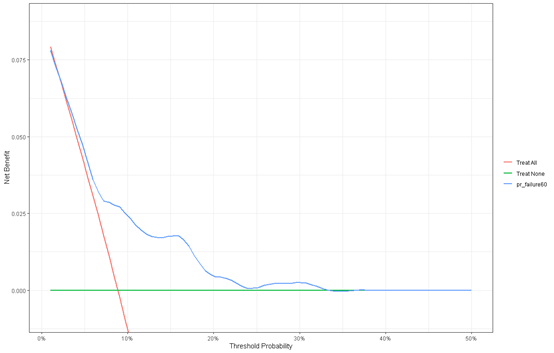
10年生存率的决策曲线
bc$pr_failure120= c(1- (summary(survfit(f1, newdata=bc), times=120)$surv))
dca(Surv(time,status) ~ pr_failure120,
data = bc,
time = 120,
thresholds = 1:50 / 100) %>%
plot(smooth = T)
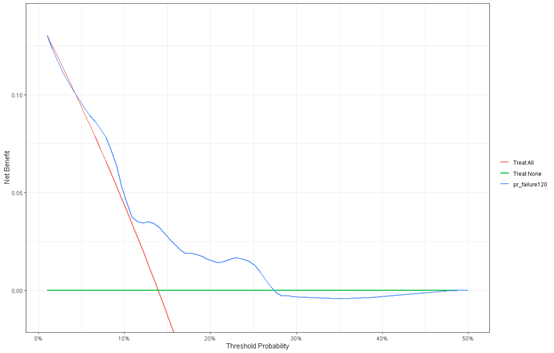
做出来的图形和stata做出来的一模一样。
还有很多功能,比如修改阈值,修改颜色和比例坐标等等,就不一一介绍了,留给大家摸索吧。

最后
以上就是紧张寒风最近收集整理的关于R语言基于dcurves包绘制COX回归临床决策曲线的全部内容,更多相关R语言基于dcurves包绘制COX回归临床决策曲线内容请搜索靠谱客的其他文章。








发表评论 取消回复