BioPython简介
Biopython工程是一个使用Python来开发计算分子生物学工具的国际团体。(http://www.python.org) Python是一种面向对象的、解释型的、灵活的语言,在计算机科学中日益流行。Python易学,语法明晰,并且能很容易的使用以C,C++或 者FORTRAN编写的模块实现扩展。
Biopython官网(http://www.biopython.org)为使用和研究生物信息学的开发者提供了一个在线的 资源库,包括模块、脚本以及一些基于Python的软件的网站链接。一般来讲,Biopython致力于通过创造高质量的和可重复利用的模块及 类,从而使得Python在生物信息学中的应用变得更加容易。Biopython的特点包括解析各种生物信息学格式的文件(BLAST, Clustalw, FASTA, Genbank...),访问在线的服务器(NCBI,Expasy...),常见和不那么常见程序的接口(Clustalw, DSSP,MSMS...),标准的序列类,各 种收集的模块,KD树数据结构等等,还有一些文档。
BioPython主要功能
- 将生物信息学文件解析为Python可用的数据结构,包含以下支持的格式:
- Blast输出结果 – standalone和在线Blast
- Clustalw
- FASTA
- GenBank
- PubMed和Medline
- ExPASy文件, 如Enzyme和Prosite
- SCOP, 包括‘dom’和‘lin’文件
- UniGene
- SwissProt
- 被支持格式的文件可以通过记录来重复或者通过字典界面来索引。
- 处理常见的生物信息学在线数据库的代码:
- NCBI – Blast, Entrez和PubMed服务
- ExPASy – Swiss-Prot和Prosite条目, 包括Prosite搜索
- 常见生物信息学程序的接口,例如:
- NCBI的Standalone Blast
- Clustalw比对程序
- EMBOSS命令行工具
- 一个能处理序列、ID和序列特征的标准序列类。
- 对序列实现常规操作的工具,如翻译,转录和权重计算。
- 利用k最近邻接、Bayes或SVM对数据进行分类的代码。
- 处理比对的代码,包括创建和处理替换矩阵的标准方法。
- 分发并行任务到不同进程的代码。
- 实现序列的基本操作,翻译以及BLAST等功能的GUI程序。
- 使用这些模块的详细文档和帮助,包括此文件,在线的wiki文档,网站和邮件列表。
- 整合BioSQL,一个也被BioPerl和BioJava支持的数据库架构。
BioPython安装:通过pip安装
(安装之前确定安装了anaconda或者miniconda或者pip)
pip install biopython测试安装
import Bio
入门小实例
#!/usr/bin/env python3
from Bio.Seq import Seq
#create a sequence object
my_seq = Seq('CATGTAGACTAG')
#print out some details about it
print ('seq %s is %i bases long' % (my_seq, len(my_seq)))
print ('reverse complement is %s' % my_seq.reverse_complement())
print ('protein translation is %s' % my_seq.translate())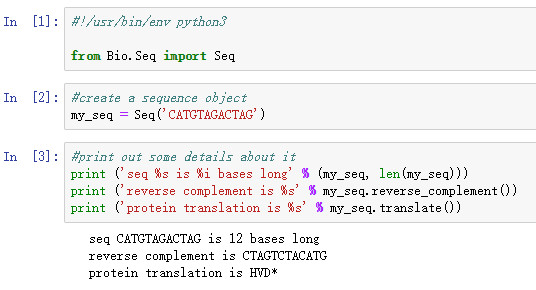
参考资料
https://biopython.org/
https://biopython.org/wiki/Download
https://github.com/biopython
最后
以上就是贪玩小懒猪最近收集整理的关于BioPython安装与入门的全部内容,更多相关BioPython安装与入门内容请搜索靠谱客的其他文章。
本图文内容来源于网友提供,作为学习参考使用,或来自网络收集整理,版权属于原作者所有。








发表评论 取消回复