pypbc的使用与一个密码方案的实现
- 一、pypbc的安装
- 二、包的相关情况
- 1、初始化
- 2、函数介绍
- 三、实现的一个方案
一、pypbc的安装
具体的安装过程可以见文章安装点这里。
讲一点自己的感受:安装其实比较简单,但对于不常使用linux的人来说,多少有一点困难,我是在deepin系统里安装的,但其实还是建议安装在ubuntu下;安装gmp、pbc等必须的依赖包的时候,可以先下载,再解压,然后运行什么./configure、make、make check、make install等相关操作,这个并不难,不过,我现在依然不是很熟练,慢慢来,总会安装好的。最近又试了一下在ubuntu下安装,好像是得输入sudo ldconfig才会安装成功。
二、包的相关情况
简单的使用可以参见github上的test.py文档。里面对于双线性对操作的几个函数如何使用演示的比较详细,耐心的一点,会看懂的。
可以进行如下操作,查看包的帮助信息,内容很全很多。
打开终端输入:python3
而后引入包:import pypbc
最后输入:help(pypbc)
在pypbc包中,有三种对象:Element,Pairing,Parameters。我们按照使用顺序来简单介绍一下。
1、初始化
首先,要对参数进行初始化,设定使用什么样的曲线,在pbc当中实现了好几种,有a.param,b.param……,在pbc里面,注意,不是在pypbc,是安装pbc的时候下载的pbc的包里面有一个param的文件夹,可以打开看看,就是一堆字母数字,都是格式化的东西。具体使用的时候有三种参数初始化的方式:
| Parameters(param_string=s) -> a set of parameters built according to s.
| Parameters(n=x, short=True/False) -> a type A1 or F curve.
| Parameters(qbits=q, rbits=r, short=True/False) -> type A or E curve.
这些参数如何设置,请参照pbc的使用文档,这对我来说有点难,不过并不妨碍使用。
例1:
#随机取两个256位的素数
q_1 = get_random_prime(256)
q_2 = get_random_prime(256)
#使用的是pbc中的a1_param参数,详见pbc_manul手册中的说明
params = Parameters( n = q_1 * q_2 )
#实例化双线性对对象,也就是经常表示的e(a,b),先这么理解吧。
pairing = Pairing( params )
这就基本完成了初始化了。
例2:
#这里的内容就可以换成pbc中param文件夹下的几种曲线参数了,但是“”“是要保留的哦。
stored_params = """type a
q 8780710799663312522437781984754049815806883199414208211028653399266475630880222957078625179422662221423155858769582317459277713367317481324925129998224791
h 12016012264891146079388821366740534204802954401251311822919615131047207289359704531102844802183906537786776
r 730750818665451621361119245571504901405976559617
exp2 159
exp1 107
sign1 1
sign0 1
"""
params = Parameters(param_string=stored_params) # type a
pairing = Pairing(params)
例3:
params = Parameters(qbits=512, rbits=160) # type a
pairing = Pairing(params)
2、函数介绍
初始化完成后,就可以做运算了,对我用到的几个函数或操作介绍一下。
先说一下,在上面提到了pypbc包的三个对象的两个,其实最重要的是Element,但大多数的操作也都与之相关,就放到这讲了;当然还有什么G1,G2,GT,Zr等相关知识,自行学习吧,我要实现的方案是基于对称的双线性群,所以并没有了解相关的非对称双线性群相关情况,也不多介绍。
#从G2中取一个随机数,并初始化一个元素,一般是取g的,也就是生成元。
g = Element.random( pairing, G2 )
#初始化一个GT元素
a = Element( pairing, GT )
#初始化一个G2元素
b = Element( pairing, G2 )
#初始化一个Zr元素
c = Element( pairing, Zr )
#初始化一个GT中的元素并置为1
Element.one( pairing, GT )
#初始化一个Zr中的元素并置为0
Element.zero( pairing, Zr )
#希望使用过程中元素的运算或赋值都使用这样的方式来进行,pairing是一定要写的,G2代表返回值的类型,
#value=就是值等于多少,也是一定要写的,G2中的元素做底数,Zr中的元素做指数,这个千万不能错。
#其实也能使用b = g ** c是同样的效果,但下面这样写更加工整,看着更明白,减少出错。
b = Element( pairing, G2, value = g ** c ) #b = g^c
注意,对运算就不能像上面那样了,得这样算,a = e(g,g),注意:type(a) = GT。
a = pairing.apply(g,g)
#哈希函数,没什么好讲的。Creates an Element from the given hash value.
hash_value = Element.from_hash( pairing, Zr, "hashofmessage”)
就这些吧,有疑问的可以自己学习,或者联系我一起学习一下。
三、实现的一个方案
方案的细节请自行去下载论文:Efficient and Privacy-preserving Online Fingerprint Authentication Scheme Over Outsourced Data或隐私保护的在线指纹认证研究与实现。

以上是方案的一些参数。

模板加密过程,其中
x
i
′
=
x
i
+
H
2
(
k
i
+
c
S
)
x' _i= x_i+H_2(k_i+c_S)
xi′=xi+H2(ki+cS),跟下面
y
y
y的处理方式一样。

生成Bloom过滤器。

生成查询。
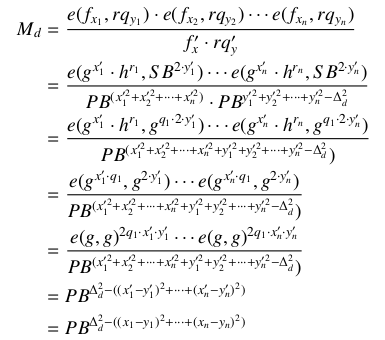
模板匹配。
虽然不是很详细,但对照下面的代码应该能看懂一二吧。
代码奉上:
#! usr/bin/env python3
# -*- coding: utf-8 -*-
from pypbc import *
from BloomFilter import *
from TimeCount import *
# system params generation
# q_1 * q_2 = n, n is the order of the group
q_1 = get_random_prime(60)
q_2 = get_random_prime(60)
params = Parameters( n = q_1 * q_2 ) # 使用的是pbc中的a1_param参数,详见pbc_manul手册中的说明
pairing = Pairing( params )
# print(params)
# 生成g,u,和h,参考e-Finga的论文。
g = Element.random( pairing, G2 )
u = Element.random( pairing, G2 )
h = Element( pairing, G2, value = u **q_2 )
SB = Element( pairing, G2, value = g ** q_1 )
PB = Element( pairing, GT )
PB = pairing.apply( g, g )
PB = Element( pairing, GT, value = PB ** q_1 )
# 哈希就在这凑合一下吧,是那么个意思,对效率的影响不大,等以后有时间了再加进来吧。
hash_value = Element.from_hash( pairing, Zr, "hashof ki + cs")
# 构造布隆过滤器,这个可是费老劲了!
Delta_d = 20
BF = BloomFilter( 8, 65536 )
Delta_d_2 = Delta_d * Delta_d
for i in range( Delta_d_2 + 1 ) :
i_value = Element( pairing, Zr, value = i )
BF.insert( Element( pairing, GT, value = PB ** i_value ) )
# Template Generation,细节看论文。
def Template_Gen( BioCode ) :
n = len( BioCode )
x = []
r = []
for i in range( n ) :
x.append( Element( pairing, Zr, value = Element( pairing, Zr, value = BioCode[i] ) + hash_value ) )
r.append( Element.random( pairing, Zr ) )
f_x = []
for i in range( n ) :
f_x.append( Element( pairing, G2, value = (g**x[i])*(h**r[i]) ) )
temp1 = Element.zero( pairing, Zr )
for i in range( n ) :
temp1 = Element( pairing, Zr, value = temp1 + x[i]**2 )
f_x_PB = Element( pairing, GT, value = PB ** (-temp1) ) # 这里说明一下,才疏学浅,就用负的幂来代替后面的除了,不知道GT群上咋求逆,反正结果是一样的。
T_u = ( f_x, f_x_PB )
return T_u
# Query Generation
def Query_Gen( BioCode ) :
n = len( BioCode )
y = []
for i in range( n ) :
y.append( Element( pairing, Zr, value = Element( pairing, Zr, value = BioCode[i] ) + hash_value ) )
q_y = []
for i in range( n ) :
temp = Element( pairing, Zr, value = y[i] + y[i] )
q_y.append( SB ** temp )
temp1 = Element.zero( pairing, Zr )
for i in range( n ) :
temp1 = Element( pairing, Zr, value = temp1 + y[i]**2 )
temp1 = Element( pairing, Zr, value = Element( pairing, Zr, value = Delta_d_2 ) - temp1 ) # 同上,结果一样,暂时不深入研究了。
q_y_PB = Element( pairing, GT, value = PB ** temp1 )
Q_u = ( q_y, q_y_PB )
return Q_u
# Match data
def Match_Data( T_u, Q_u ) :
f_x = T_u[0]
f_x_PB = T_u[1]
q_y = Q_u[0]
q_y_PB = Q_u[1]
n = len( f_x )
# 感觉这个e_list的说法怪怪的,感觉应该是这么用。
e_list = []
for i in range( n ) :
e_list.append( Element( pairing, GT ) )
for i in range( n ) :
e_list[i] = pairing.apply( f_x[i], q_y[i] )
up_value= Element.one( pairing, GT )
for i in range( n ) :
up_value = Element( pairing, GT, value = up_value * e_list[i] )
down_value = Element( pairing, GT, value = f_x_PB * q_y_PB )
M_d = Element( pairing, GT, value = up_value * down_value)
return M_d
# Test Fuction,小小的设计一下测试。
def test( dims, times ) :
t_TG = timing( Template_Gen, 1 )
t_QG = timing( Query_Gen, 1 )
t_MD = timing( Match_Data, 1 )
clocktime_TG_sum = 0
clocktime_QG_sum = 0
clocktime_MD_sum = 0y
Result_list = []
for i in range( times ) :
# 尽可能的减少人为的因素,BioCode码是随机的。
BC_1 = []
for i in range( dims ) :
BC_1.append( get_random(255) )
# 加一些扰乱因子,算是每次采集信息会有一点点不同的样子,但好像又有一点点小问题,也没有想像中的那么美好。
BC_2 = []
for i in range( dims ) :
BC_2.append( BC_1[i] + get_random( 3 ) )
T_u, clocktime_TG = t_TG( BC_1 )
clocktime_TG_sum += clocktime_TG
Q_u, clocktime_QG = t_QG( BC_2 )
clocktime_QG_sum += clocktime_QG
M_d, clocktime_MD = t_MD( T_u, Q_u )
clocktime_MD_sum += clocktime_MD
Result_list.append( BF.is_exist( M_d ) )
return ( clocktime_TG_sum, clocktime_QG_sum, clocktime_MD_sum, Result_list )
if __name__ == "__main__":
test_1 = test( 2, 5 )
print( test_1[0] )
print( test_1[1] )
print( test_1[2] )
# print( test_1[3] )
以上是代码主体,实现了论文方案的主要算法,下面是需要引入的两个包,也是我写的,挺垃圾,不过放在这大家一起改进一下。
布隆过滤器,得安装numpy,用的时候文档名要写成BloomFilter.py:
#! usr/bin/python3
# -*- coding: utf-8 -*-
import numpy as np
import hashlib
class BloomFilter(object) :
def __init__( self, times, dims ) :
self.times = times
self.dims = dims
self.judge = np.zeros( (self.dims,), dtype = np.int )
def insert( self, item ) :
for i in range(self.times) :
h_values = self.hash_( item * i )
for h_value in h_values :
self.judge[ h_value ] = 1
def is_exist( self, item ) :
flag = True
for i in range(self.times) :
h_values = self.hash_( item * i )
for h_value in h_values :
if self.judge[h_value] == 0 :
flag = False
break
if flag == False :
break
return flag
def hash_( self, message ) :
temp = hashlib.sha512()
temp.update( str( message ).encode('utf-8') )
temp1 = temp.hexdigest()
ret = []
for i in range(5) :
ret.append( int(temp1[ i * 4 : (i+1) * 4 ],16) )
return ret
if __name__ == "__main__":
example = [ 255, 232, 323, 434, 329, 213 ]
BF = BloomFilter( 8, 65536 )
for exam in example :
BF.insert( exam )
Result = BF.is_exist(255)y
print(Result)
计时函数,用法同上,得跟第一个文档中写的名字对应起来。
#! usr/bin/env python3
# -*- coding: utf-8 -*-
import time
# time-counting function
# reference : github/paillier-gmpy2
def timing(f, c = 0):
def wrap(*args):
time1 = time.time()
return_value = f(*args)
time2 = time.time()
clock_time = time2 - time1
if c == 0:
return return_value
else :
return return_value, clock_time
return wrap
若有侵权,请联系yuezelun@163.com。
若有需要,也请联系yuezelun@163.com。
若转载,请注明出处,尤其是代码,虽写的不咋的,但费了不少劲呢!
参考文献:
1.Zhu, H., Wei, Q., Yang, X., Lu, R., Li, H.: Efficient and privacy-preserving online
fingerprint authentication scheme over outsourced data. IEEE Transactions on
Cloud Computing pp. (2018). https://doi.org/10.1109/TCC.2018.2866405
2.魏晴. 隐私保护的在线指纹认证研究与实现[D].西安电子科技大学,2018.
最后
以上就是温婉白昼最近收集整理的关于pypbc的使用与一个密码方案的实现一、pypbc的安装二、包的相关情况三、实现的一个方案的全部内容,更多相关pypbc内容请搜索靠谱客的其他文章。








发表评论 取消回复