上一篇文章中使用 Pavia 大学的数据进行了数据探索性分析和可视化。可视化的代码,在上一篇文章中已经给出,只需要修改数据即可实现相应的可视化。本文重点介绍数据的降维和特征选择。
文章目录
- Github 源码获取
- Dataset:Indian Pines
- Python读取.mat 格式数据
- 加载数据
- 图像的真实标签信息
- 将图像数据转换为CSV存储
- PCA降维
- 可视化PCA之后的光谱
- SVM分类
- 可视化分类结果
- Reference
Github 源码获取
https://github.com/datamonday/HSI-Analysis
Dataset:Indian Pines
下载地址:Link
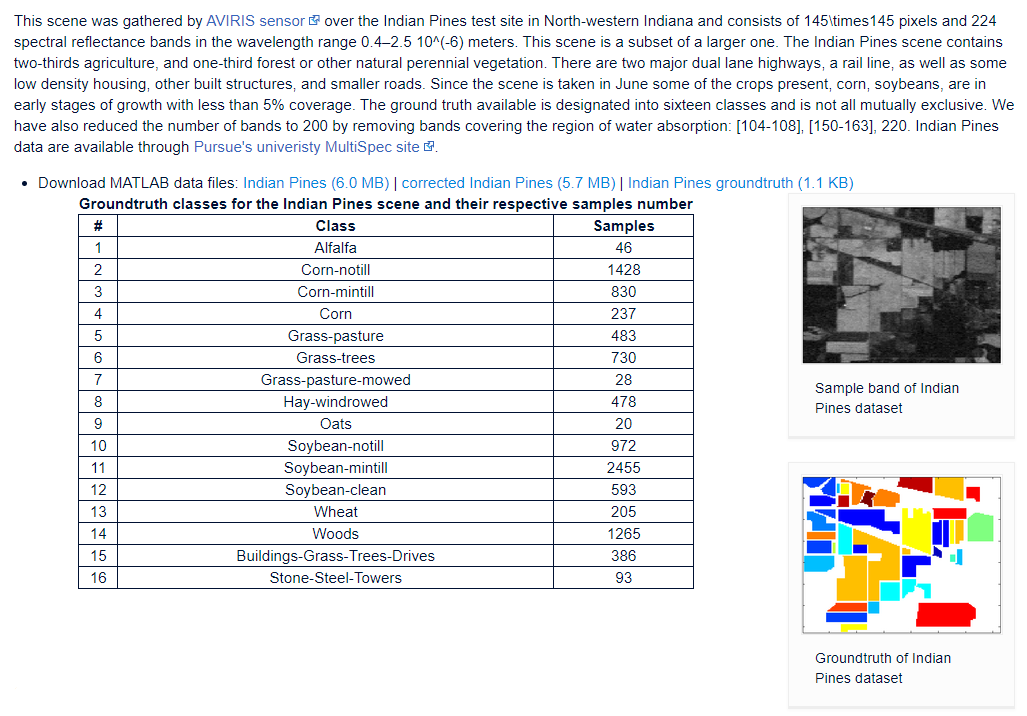
易知,该数据集包含16类不同的对象,其具体的标签如上图中的表格所示。每张图像的尺寸为 145×145像素。
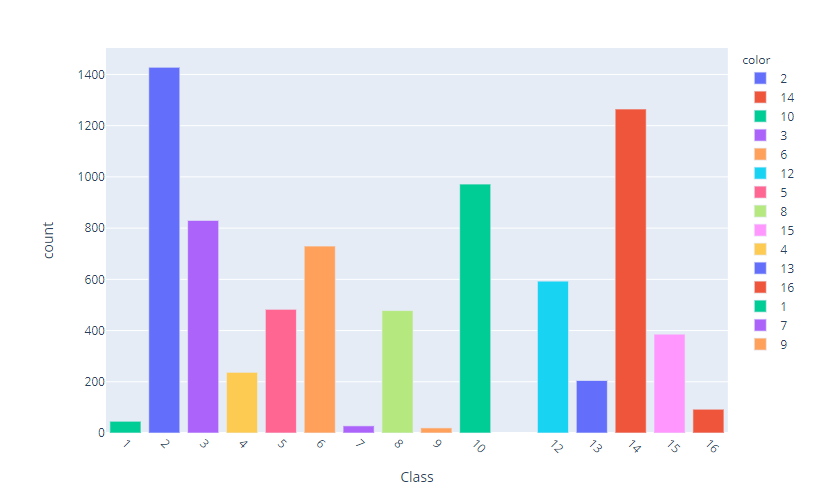
个类别标签柱状图:
Python读取.mat 格式数据
import numpy as np
import pandas as pd
import seaborn as sns
import matplotlib.pyplot as plt
from sklearn.preprocessing import StandardScaler
from sklearn.model_selection import train_test_split
from sklearn.svm import SVC
from sklearn.metrics import accuracy_score, classification_report, confusion_matrix
from scipy.io import loadmat
indian_pines = loadmat('dataset/Indian_pines_corrected.mat')
# loadmat方法加载数据后会返回一个Python字典的数据结构
indian_pines.keys()
dict_keys(['__header__', '__version__', '__globals__', 'indian_pines_corrected'])
# 由关键字可知,读取'indian_pines_corrected'字段
indian_pines = indian_pines['indian_pines_corrected']
# 查看数据的shape
indian_pines.shape
(145, 145, 200)
# 查看数据的类型,numpy.ndarray类型
type(indian_pines)

# 加载像素标签数据
indian_pines_gt = loadmat('dataset/Indian_pines_gt.mat')
print(indian_pines_gt.keys())
dict_keys(['__header__', '__version__', '__globals__', 'indian_pines_gt'])
indian_pines_gt = indian_pines_gt['indian_pines_gt']
indian_pines_gt.shape
(145, 145)
由像素标签的维度和高光谱图像数据可知,光谱数据中,每个像素对应一个类别,并且在不同波段的图像中相同位置的像素所属的类别相同!
查看样本中的标签:

注意,标签0表示没有关键的类别,是无用的数据,所以在可视化的时候需要将这一维度去掉。
加载数据
X = indian_pines
y = indian_pines_gt
print(f"X shape: {X.shape}ny shape: {y.shape}")
sns.axes_style('whitegrid')
fig = plt.figure(figsize=(12, 6))
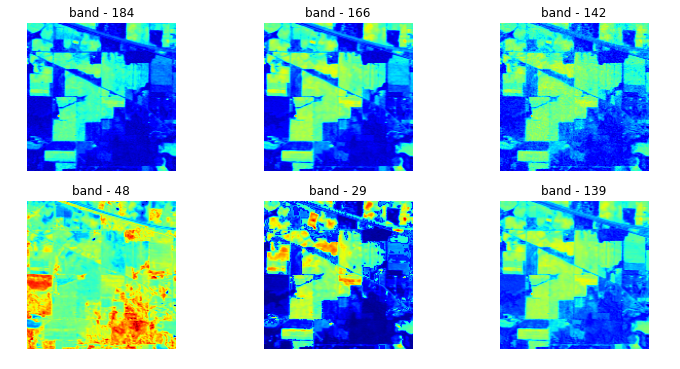
# 不同光谱下的图像示例
for i in range(1, 1+6):
fig.add_subplot(2, 3, i)
q = np.random.randint(X.shape[2])
plt.imshow(X[:, :, q], cmap='jet')
plt.axis('off')
plt.title(f'band - {q}')
图像的真实标签信息
!pip install plotly
import plotly.express as px
cls = px.imshow(y, color_continuous_scale='jet')
cls.update_layout(title='Ground Truth', coloraxis_showscale=True)
cls.update_xaxes(showticklabels=False)
cls.update_yaxes(showticklabels=False)
cls.show()

将图像数据转换为CSV存储
def extract_pixels(X, y, save_name='indian_pines_all'):
q = X.reshape(-1, X.shape[2])
df = pd.DataFrame(q)
df = pd.concat([df, pd.DataFrame(y.ravel())], axis=1)
df.columns= [f'band{i}' for i in range(1, 1+X.shape[2])]+['class']
df.to_csv(f'dataset/{save_name}.csv')
return df
df = extract_pixels(X, y, save_name='indian_pines_all')
df
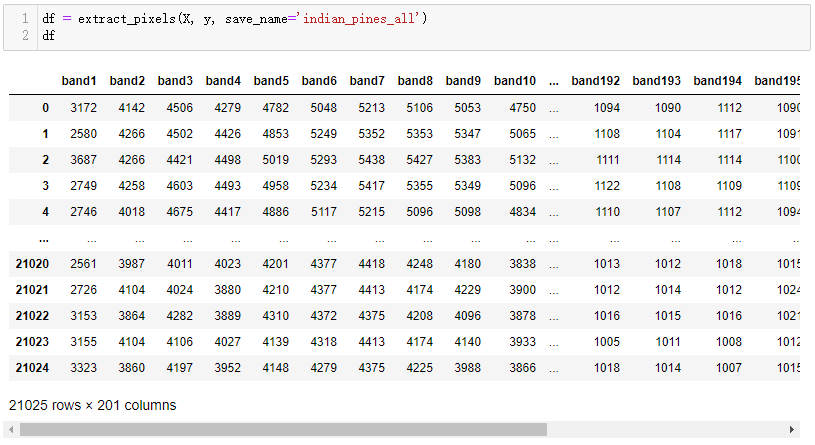
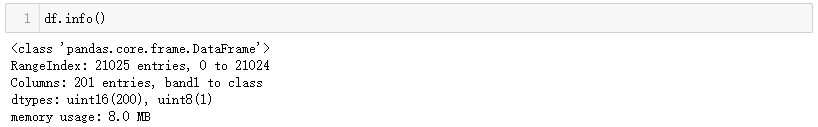
查看转换后的数据信息
PCA降维
降维(Dimensionality Reduction, DR)用于减少数据的维数,降低计算成本。有助于提高高光谱图像(HSI)像素分类的准确性。
特征选择是选择数据集特征维的过程,该维有助于机器学习任务的模式,例如分类,聚类等。 这可以通过使用不同的方法来实现,例如相关分析,单变量分析等。
降维可以采用两种类型:
- 特征选择:选择数据集特征维的过程,可以通过使用不同的方法来实现,例如相关分析,单变量分析等。
- 特征提取:通过选择和/或组合现有特征以创建缩小的特征空间来查找新特征的过程,同时仍能准确,完整地描述数据集而不会丢失信息。
基于准则函数和收敛过程,降维技术也分为凸(Convex)和非凸(Non-Convex)。 一些流行的降维技术包括PCA,ICA,LDA,GDA,KernelPCA,Isomap,局部线性嵌入(Local linear embedding,LLE),Hessian LLE等。
本文使用主成分分析(PCA)来减少数据的维数。
from sklearn.decomposition import PCA
pca_components = 40
pca = PCA(n_components = pca_components)
dt = pca.fit_transform(df.iloc[:, :-1].values)
q = pd.concat([pd.DataFrame(data = dt), pd.DataFrame(data = y.ravel())], axis = 1)
q.columns = [f'PC-{i}' for i in range(1, pca_components+1)]+['class']
q.head()
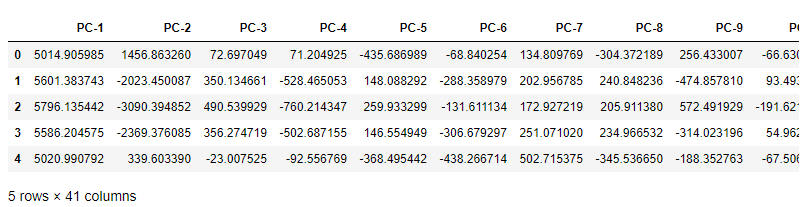
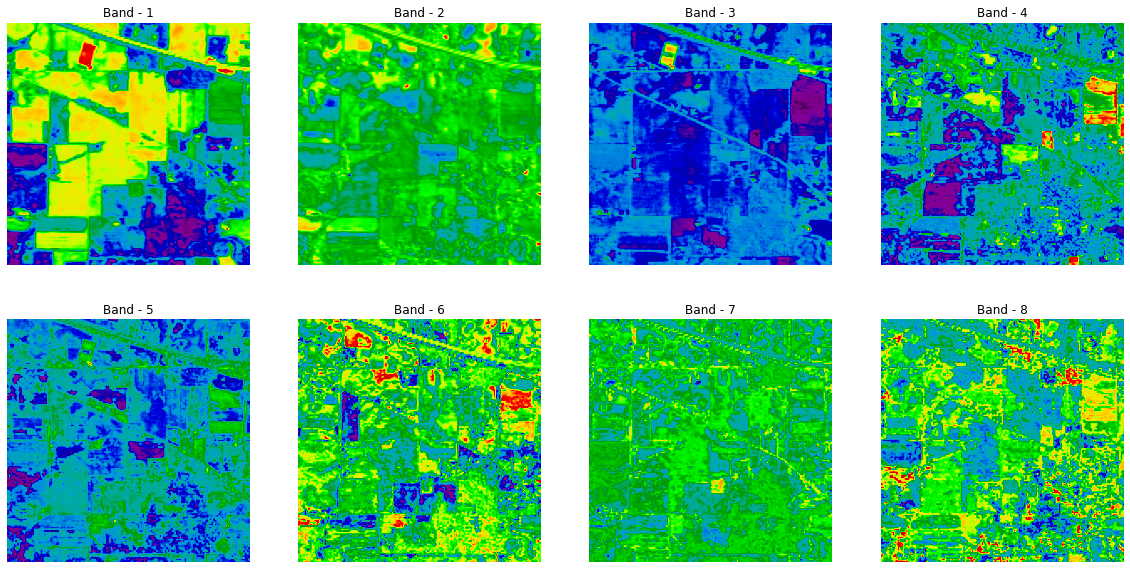
可视化PCA之后的光谱
fig = plt.figure(figsize = (20, 10))
for i in range(1, 1+8):
fig.add_subplot(2,4, i)
plt.imshow(q.loc[:, f'PC-{i}'].values.reshape(145, 145), cmap='nipy_spectral')
plt.axis('off')
plt.title(f'Band - {i}')
plt.savefig('IP_PCA_Bands.png')
SVM分类
使用经过PCA降维后的数据进行分类。
分类是指预测建模问题,其中针对给定的输入数据预测了类别标签。分类可分为:
- Classification Predictive Modeling
- Binary Classification
- Multi-Class Classification
- Multi-Label Classification
- Imbalanced Classification
我们正在处理“多类分类”问题。高光谱图像(HSI)的分类有不同的分类算法,例如:
- K-Nearest Neighbors
- Support Vector Machine
- Spectral Angle Mapper
- Convolutional Neural Networks
- Decision Trees e.t.c
本文使用支持向量机(SVM)对高光谱图像(HSI)进行分类。
x = q[q['class'] != 0]
X = x.iloc[:, :-1].values
y = x.loc[:, 'class'].values
names = ['Alfalfa', 'Corn-notill', 'Corn-mintill', 'Corn', 'Grass-pasture', 'Grass-trees',
'Grass-pasture-mowed', 'Hay-windrowed', 'Oats', 'Soybean-notill', 'Soybean-mintill',
'Soybean-clean', 'Wheat', 'Woods', 'Buildings Grass Trees Drives', 'Stone Steel Towers']
X_train, X_test, y_train, y_test = train_test_split(X, y, test_size=0.20, random_state=11, stratify=y)
svm = SVC(C=100, kernel='rbf', cache_size=10*1024)
svm.fit(X_train, y_train)
ypred = svm.predict(X_test)
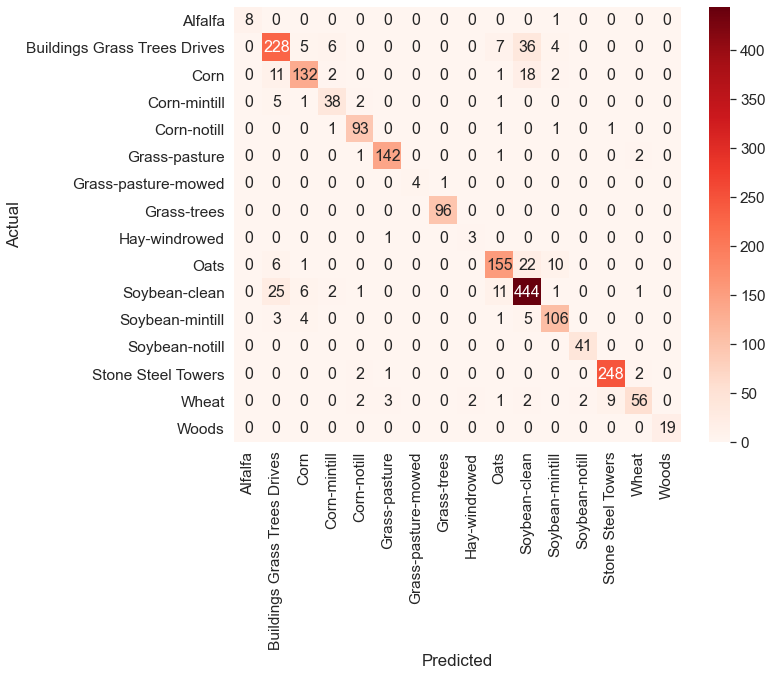
data = confusion_matrix(y_test, ypred)
df_cm = pd.DataFrame(data, columns=np.unique(names), index = np.unique(names))
df_cm.index.name = 'Actual'
df_cm.columns.name = 'Predicted'
plt.figure(figsize = (10,8))
sns.set(font_scale=1.4)#for label size
sns.heatmap(df_cm, cmap="Reds", annot=True,annot_kws={"size": 16}, fmt='d')
plt.savefig('cmap.png', dpi=300)
查看测试集的(20%)分类混淆矩阵:
查看分类评价指标:
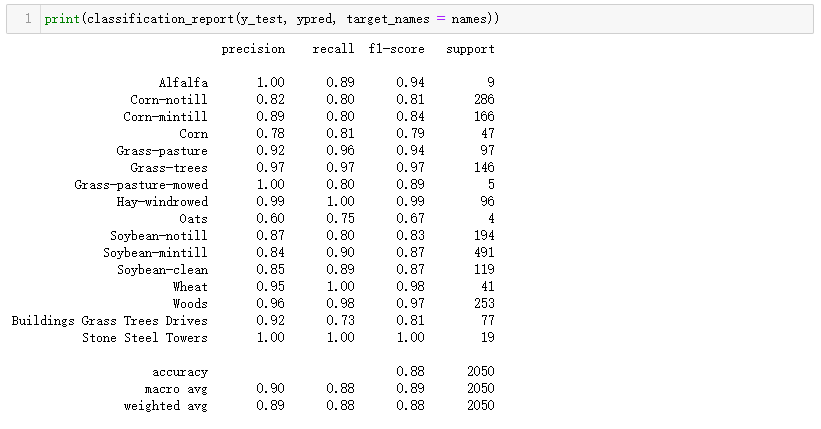
有些类别的分类结果不准确,可能与其样本数量有关,也可能是因为不同类别之间的差距不明显。
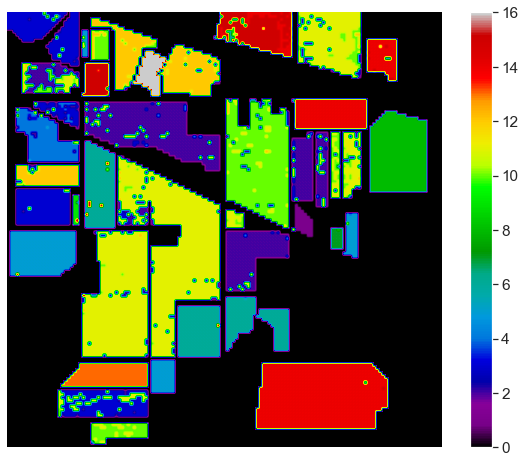
可视化分类结果
# Classification Map
l=[]
for i in range(q.shape[0]):
if q.iloc[i, -1] == 0:
l.append(0)
else:
l.append(svm.predict(q.iloc[i, :-1].values.reshape(1, -1)))
clmap = np.array(l).reshape(145, 145).astype('float')
plt.figure(figsize=(10, 8))
plt.imshow(clmap, cmap='nipy_spectral')
plt.colorbar()
plt.axis('off')
plt.savefig('IP_cmap.png')
plt.show()
绘制真实数据标签:
# 真实数据
plt.figure(figsize=(10, 8))
plt.imshow(y, cmap='nipy_spectral')
plt.colorbar()
plt.axis('off')
plt.savefig('IP_cmap.png')
plt.show()
真实标签与SVM分类结果对比:
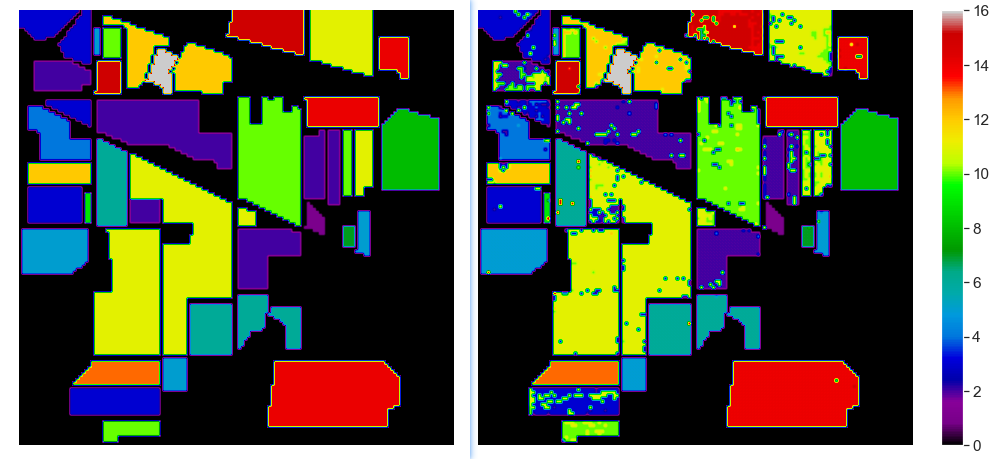
可以看出,目前结果还是很不错的,虽然有些噪点。只是使用默认参数取得上图的结果还是可以接收的,后续通过一些超参数优化方法,数据预处理步骤可能获得更好的结果。
Reference
Dimensionality Reduction in Hyperspectral Images using Python
Hyperspectral Image Analysis — Classification
最后
以上就是从容红牛最近收集整理的关于高光谱图像分析:分类 IIGithub 源码获取Dataset:Indian PinesPCA降维SVM分类Reference的全部内容,更多相关高光谱图像分析:分类内容请搜索靠谱客的其他文章。








发表评论 取消回复