(1)细胞样本名字的标准化
细胞名字要求:“样本号” + “-” + “细胞号”
例:

注:上面的是原来的名字,我们要保留subject和cell,前面的SCRNA和TEMRA不要
library(stringr)
head(colnames(d1))
Name <- colnames(d1)
Name_1 <- str_replace(Name, "SCRNA_", "")
Name_2 <- str_replace(Name_1, "TEMRA_", "")
head(Name_2)
colnames(d1) <- Name_2
注:用 str.replace将两个多余的部分去掉
(2)基因名字是ENSYMBL还是symbool
例:

注:上面的基因名字不是symbol,而是带版本号的ENSYMBL,需要去版本号,然后转化
d1 <- File
library(stringr)
d1$id <- unlist(str_split(d1$id,"[.]",simplify=T))[,1]
d1[,1][1:5]
注:上面这是去掉版本号
library(limma)#avereps所在的函数包
data <- d1
#将基因ENSEMBL转化为geneSymbol
library(AnnotationDbi)
library(clusterProfiler)
library(org.Hs.eg.db)
keytypes(org.Hs.eg.db)
gene.ens.id <- data[,1]
genes <- AnnotationDbi::select( org.Hs.eg.db, keytype = "ENSEMBL", keys = gene.ens.id, columns = c("SYMBOL"))
name_data <- data[,1]
name_data_gai <- genes[match(name_data, genes$ENSEMBL),]
data <- as.matrix(data)
rownames(data) <- name_data_gai[,2]
exp <- data[,2:ncol(data)]
dimnames <- list(rownames(exp),colnames(exp))
data <- matrix(as.numeric(as.matrix(exp)),nrow=nrow(exp),dimnames=dimnames)
data <- avereps(data)
#因为在这里很容易出现空白名字,所以,得去掉,不是空白名字,而是名字NA,有一个
data <- data[-match(NA,rownames(data)),]
注:这就将名字改过来了
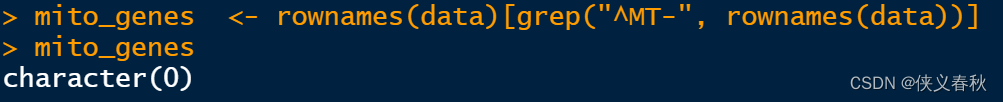
注:但注意:线粒体基因没有MT-开头,所以在之后的计算线粒体基因活性的步骤中,这一步将被过滤,当然,其实对实验的影响很小,如果之后有更好的方法来替换gene id又能保留MT-,将更新。
(3)细胞线粒体基因前面是MT还是MT-还是说啥前缀也没有,还有一点,老鼠是mt-
mito_genes <- rownames(testdf)[grep("^MT-", rownames(testdf))]
mito_genes #线粒体基因
注:这个代码是用来检测线粒体基因的开头是啥的,又是后要注意,有些情况下,作者的线粒体基因以MT开头,在后面同样不能筛选线粒体基因,为啥呢?因为还有很多无辜的基因会被你一同算进去。这里的testdf是一开始输入的原始数据。如果基因名字不是行名而是第一列,请更改代码:rownames(testdf) 为 testdf[,1]
注意:有些是MT_,坑得很
(4)细胞到底是 counts 还是 标准化以后的数据
(1)按照正常流程录入的初始数据因该是原始counts数据,它会存在test.seu@count中;然后对原始数据进行筛选,将及其不活跃的细胞和及其不活跃的细胞,还有线粒体基因表达过于高的细胞踢掉;之后在进行独有的标准化:每细胞基因的计数除以总计数,再乘以scale.factor,最后再log(n+1),其scale.factor是人为设置,一般是10000,这一步的作用是消除每一个细胞的测序深度
(2)如果线粒体基因不是以MT-开头,则,不能删除线粒体基因极度高表达的细胞
(3)如果是标准化以后数据,一般是TPM和FKTM,它已经经过缩聚处理,只需要再log就行,pbmc@data = log(TPM + 1)
### 常规流程
test.seu=CreateSeuratObject(counts = testdf)
test.seu <- NormalizeData(test.seu, normalization.method = "LogNormalize", scale.factor = 10000)
test.seu <- FindVariableFeatures(test.seu, selection.method = "vst", nfeatures = 2000)
......
### TPM数据
test.seu1=CreateSeuratObject(counts = log(mat_tpm+1))
test.seu1 <- FindVariableFeatures(test.seu1, selection.method = "mvp", nfeatures = 2000)
# 或者
test.seu=CreateSeuratObject(counts = mat_tpm)
test.seu[["RNA"]]@data=as(as.matrix(log(mat_tpm + 1)), "dgCMatrix")
test.seu <- FindVariableFeatures(test.seu, selection.method = "mvp", nfeatures = 2000)群聊:
203241137
咱不定期邀请大佬给大家讲解各个R语言技术之间的原理与步骤
最后
以上就是花痴钢笔最近收集整理的关于单细胞测序分析【1】---样本的基础处理的全部内容,更多相关单细胞测序分析【1】---样本内容请搜索靠谱客的其他文章。








发表评论 取消回复