构建数据库
mkdir bio-db上传、拷贝、移动要构建数据库的FASTA文件到数据库
上传使用sftp等工具
拷贝
cp xxxxx.fasta bio-db
移动
mv xxxxx.fasta bio-db
# 切换到bio-db 文件夹
cd bio-db构建本地数据库
# 更多makeblastdb命令,请用 makeblastdb -h 查询,这里只是举例
makeblastdb -in input_file -dbtype molecule_type -parse_seqids -out database_name -logfile File_Name
例
makeblastdb -in xxxxx.fasta -dbtype nucl -parse_seqids -out A9.fasta -logfile A9.txt
-dbtype 后接序列类型,nucl为核酸,prot为蛋白
-parse_seqids 推荐加上
-out 后接数据库名
-logfile 日志文件,如果没有默认输出到屏幕本地数据库构建成功
获取目标序列
blastn -query 参考目的片段(可内置多个参考序列) -db 数据库存放的位置 -out 输出的结果文件名
blastn -query 11.fasta -db A9.fasta -out A9blast.txt -outfmt 6
-outfmt:输出文件格式,
0:成对输出,与在线比对结果相同
5:输出XML格式
6:输出table格式
7:输出带有注释行的table格式,有以下参数可通过’’7 参数’’的形式接在-outfmt后使用:
输出结果
cat A9blast.txt结果如下图
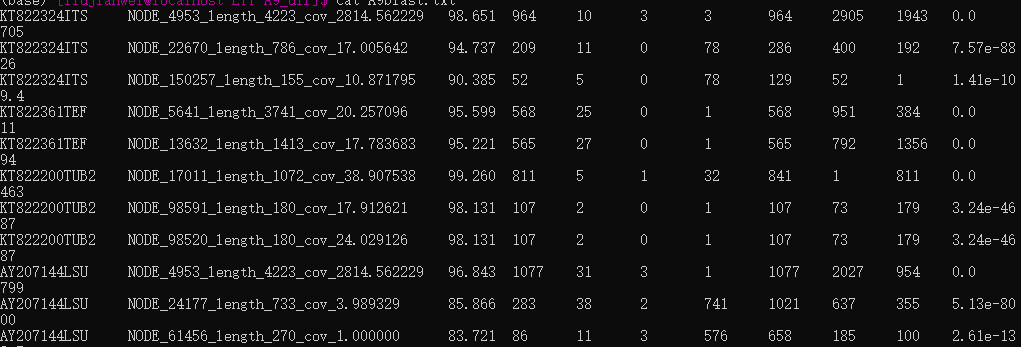
从左到右各列参数为 Query_id Subject_id %_identity alignment_length mismatches gap_openings q. start q. end s. start s. end e-value bit_score
本次测试成功获得四种类型(ITS/TEF/TUB2/LSU)基因片段
最后
以上就是精明小懒虫最近收集整理的关于Linux: 使用blastn 快速从二代数据组装基因组中同时获取多个核酸目标片段构建数据库获取目标序列输出结果 的全部内容,更多相关Linux:内容请搜索靠谱客的其他文章。
本图文内容来源于网友提供,作为学习参考使用,或来自网络收集整理,版权属于原作者所有。








发表评论 取消回复