SmileGNN:基于SMILES和GNN的药物相互作用预测
| 论文题目 | SmileGNN: Drug-Drug Interaction Prediction Based on SMILES and Graph Neural Network |
|---|---|
| 论文出自 | Research Article(Preprints),June 10th, 2021 |
论文链接:SmileGNN
文章目录
- SmileGNN:基于SMILES和GNN的药物相互作用预测
- 一、SmileGNN模型?
- 二、各模块详细介绍?
- 三、实验?
- 四、思考?
一、SmileGNN模型?
SmileGNN:利用SMILES数据构造药物的结构特征。利用图神经网络得到知识图中药物的拓扑特征。聚合药物的结构和拓扑特征以预测新药对的相互作用。(阈值0.5,以此分类为存在DDI或不存在DDI)
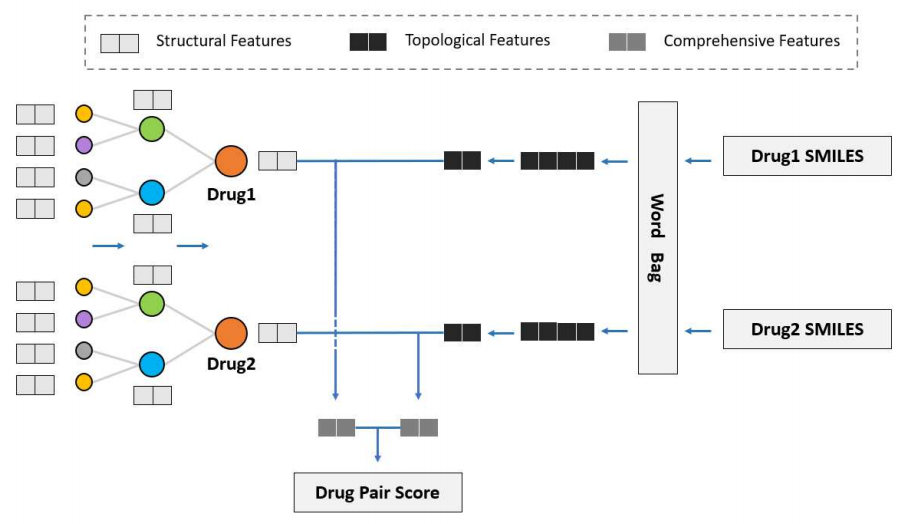
二、各模块详细介绍?
- 药物结构特征
数据库:DrugBank。
SMILES可以用一串字符描述三维化学结构,如图是药物亚叶酸及其相应SMILES的二维图形。
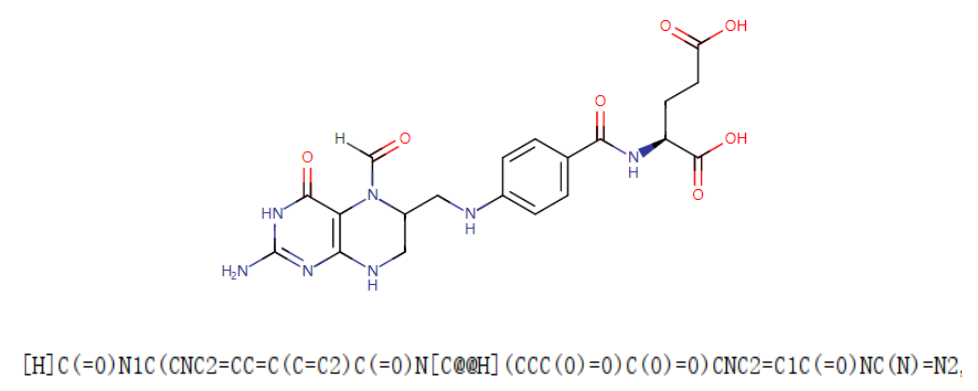
SMILES2Vec方法是将自然语言处理中的Seq2seq技术应用到SMILES字符串中,将化学结构信息作为深度神经网络的输入变量来预测化合物的物理性质。SMILES2Vec在预处理过程中删除一些长的 SMILES ,并对剩下的 SMILES进行独热编码。**将每个SMILES转换为长度为26的向量。**根据这种预处理方法,对药物的化学结构进行了预处理。
![[外链图片转存失败,源站可能有防盗链机制,建议将图片保存下来直接上传(img-tXzFiLwE-1638878663613)(D:DBLabMyNoteTyporaImagesimage-20211205194911636.png)]](https://www.shuijiaxian.com/files_image/2023062822/2e6b2de6e1a54d398c24d906dbe7d64e.png)
-
药物拓扑特征
-
KG的构造
利用两个数据库中的数据构建KG,获得相应数据库中药物的拓扑特征。
-
KEGG
-
PDD
RDF(资源描述框架)Bio2RDF项目提供了将数据转换为n-quads或其他RDF格式的工具。然后,使用RDFlib库解析这些n-quads数据,并将它们以便于后续KG生成嵌入特征的格式划分为三元组(实体、关系、实体),如图3所示。
[外链图片转存失败,源站可能有防盗链机制,建议将图片保存下来直接上传(img-XskQ0LnG-1638878663613)(D:DBLabMyNoteTyporaImagesimage-20211130102152433.png)]
引入一个度量来评估KG。密度用于描述图/网络中节点之间的边连接密度。对于有L条边和N个节点的图G,密度计算公式:
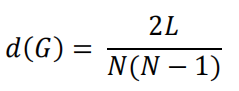 分别用KEGG和PDD构建了两个KG。
分别用KEGG和PDD构建了两个KG。
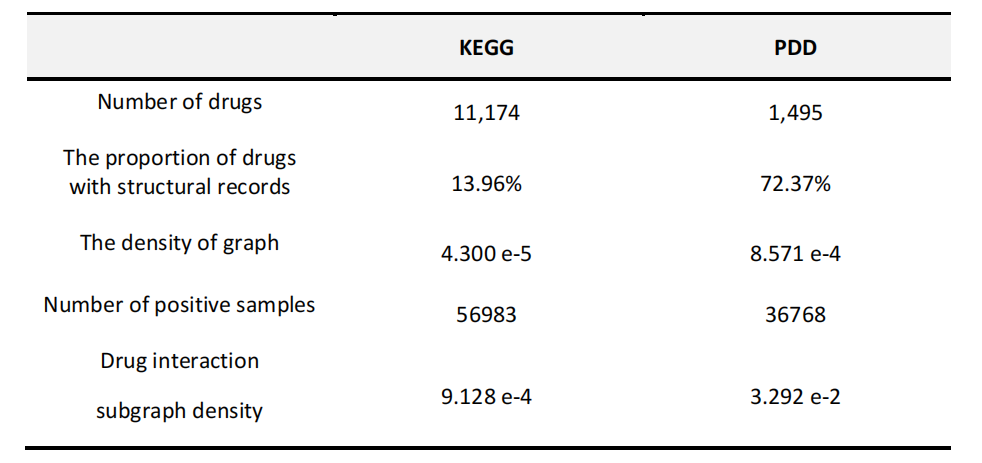 从表中可以看出,KEGG数据集中的药物种类较多,但图形本身相对稀疏,具有结构记录的药物比例相对较低。PDD数据集的药物类型较少,但图形更密集,具有结构记录的药物比例更高。
从表中可以看出,KEGG数据集中的药物种类较多,但图形本身相对稀疏,具有结构记录的药物比例相对较低。PDD数据集的药物类型较少,但图形更密集,具有结构记录的药物比例更高。 -
-
拓扑特征的提取
通常使用KG预测DDI的模型只能捕获小范围内的数据信息。**为了扩大邻居域,获取KG中丰富的实体信息,探索药物与其他实体之间的潜在相关性,提出了KGNN模型。**KGNN通过GNN提取药物的高阶结构和语义关系,通过KG图学习药物和药物邻居的表示。
对于每个实体,该模型从实体的域中提取多个实体,并聚合这些实体的信息以形成实体的拓扑特征表示。实体聚合方法有三种:和聚合是一种叠加操作,连接是一种连接操作,邻居只考虑邻居而不考虑节点本身的信息。这三种聚合方法缩写为sum、concat和neigh。
-
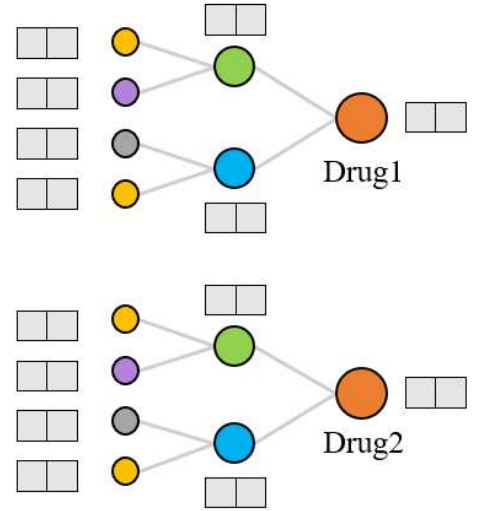
- 回顾KGNN
![[外链图片转存失败,源站可能有防盗链机制,建议将图片保存下来直接上传(img-3tRYGeAY-1638878663614)(D:DBLabMyNoteTyporaImagesimage-20211116164837801.png)]](https://www.shuijiaxian.com/files_image/2023062822/b7ba8038d12440abab31ccf3dfe6fbba.png)
- 该模型采用二分交叉损失熵作为损失函数:
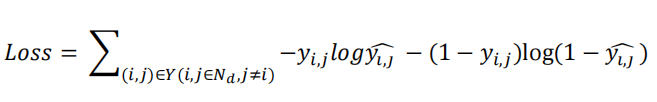
三、实验?
在本文中,DDI的预测被认为是一项二分类任务,即只需判断药物对之间是否存在可能的DDI。
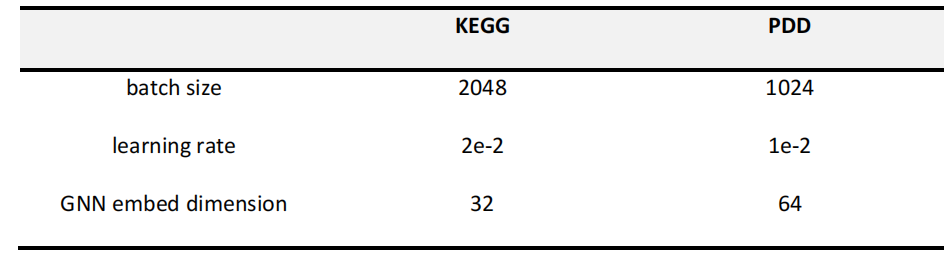
- 使用的最终参数:
- 与其他模型的比较:
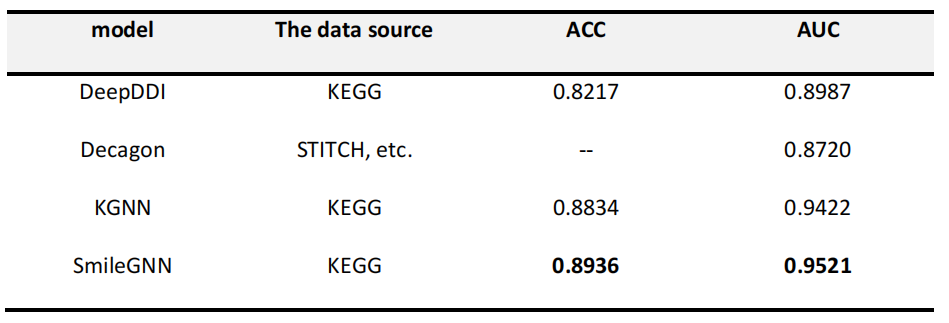
本文提出的新模型SmileGNN结合了药物的拓扑特征和结构特征,比单独使用拓扑特征的Decagon和KGNN模型或单独使用结构特征的DeepDDI模型具有更好的性能。
- 消融实验
**SmileGNN将药物结构特征的使用添加到KGNN中,并集成多源信息以预测新的DDI。**与KGNN模型原始性能的比较是一个消融实验,以比较和分析新药结构特征对模型性能的影响。
在KEGG和PDD数据集上进行实验,分别对sum、concat和neigh三种药物拓扑特征聚合类型进行实验。实验结果如表4所示。
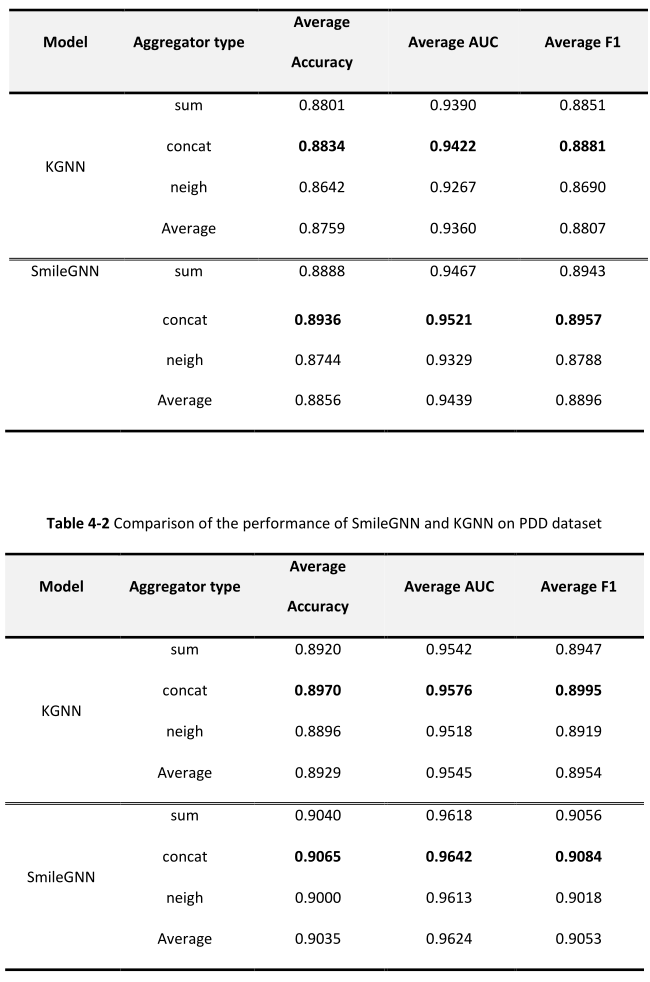
1.对于KEGG和PDD数据集,使用药物结构特征的SmileGNN在所有三种药物拓扑特征聚合方法中的性能都优于KGNN。
2.与KGNN模型一致,SmileGNN在使用concat获取药物拓扑特征时效果最好。
基本可以得出以下结论:
1.在稠密图上,从模型中学习到的药物拓扑信息更加丰富,能够更好地表示药物的拓扑特征。
2.在PDD数据中,药物结构对应的药物比例较高,药物结构特征对模型的影响较大,这是积极的。
-
结果
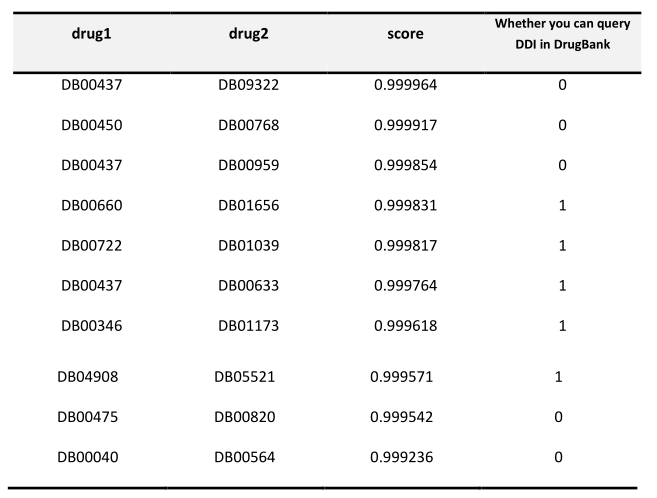
前十名预测的药物新相互作用中有五个是从最新的数据库中验证的,证明了SmileGNN的可信度。
四、思考?
SmileGNN可以很好地利用已知的药物结构信息和药物拓扑信息来预测DDI。
不足:
一方面,该模型受到药物结构信息不足的限制。
另一方面,KG中拓扑信息的学习是相对盲目和随机的。
最后
以上就是甜甜黄豆最近收集整理的关于AI医药论文解读--SmileGNN:基于SMILES和GNN的药物相互作用预测SmileGNN:基于SMILES和GNN的药物相互作用预测的全部内容,更多相关AI医药论文解读--SmileGNN:基于SMILES和GNN内容请搜索靠谱客的其他文章。








发表评论 取消回复