摘要
脱氧核糖核酸的复制是细胞中发生的最关键的任务之一,它必须得到精确的调控。这一过程始于复制起源(ORIs),因此识别这些位点对于更深入地了解与基因表达调控相关的细胞过程和功能是至关重要的。考虑到ORIs所执行的重要任务,一些实验和计算方法已经发展来预测这些地点。然而,现有的ORIs计算预测器有一定的限制,如只构建单特征编码模型,有限的系统特征工程工作,以及无法验证模型的鲁棒性。因此,我们开发了一种新的物种特异性酵母预测器,称为yORIpred,可以准确地识别酵母基因组中的ORIs。为了开发yORIpred,我们首先通过探索8种不同的基于序列的编码和5种不同的机器学习分类器,构建了最优的40个基线模型。随后,将40个模型的预测概率作为新的特征向量,并使用5种不同的分类器独立进行迭代特征学习方法。系统分析表明,与其他四种算法相比,支持向量机算法(yORIpred)学习到的特征表示可以很好地区分ORIs和非ORIs之间的分布特征。综合基准实验表明,在相同的训练数据集上,yORIpred在现有预测器上取得了优越和稳定的性能。此外,独立评价显示了yORIpred的最佳和准确的性能,从而强调了迭代特征表示的重要性。为了方便用户在不经历任何数学、统计或计算麻烦的情况下获得他们预期的结果,我们为yORIpred预测器开发了一个web服务器,可在以下网址获得:http://thegleelab.org/yORIpred
关键词:迭代特征表示支持向量机复制起源机器学习
介绍
脱氧核糖核酸(DNA)是生命遗传信息的主要载体。在细胞分裂之前,需要复制包括DNA分子的复制,以便在新的子细胞中适当分布。这种发生在分裂细胞中的DNA复制过程被称为DNA复制或半保守复制[1]。DNA复制分别发生在原核生物和真核生物的细胞质和细胞核中。然而,DNA复制的基本过程仍然是相同的。复制的起点是基因组复制的位点,因此它必须是严格调控的[2]。与真核生物基因组[4]中存在多个复制起源(ORIs)相比,细菌基因组只有一个复制起源[3]。由于DNA的复制是在ORIs中开始的,因此有必要识别这些位点,以便更好地识别遗传信息的传播。
一些体外和体内实验技术,如染色质免疫沉淀(ChIP)、芯片测序、DNaseI足迹技术、电泳迁移率测定、凝胶阻滞测定、等温滴定量热法、复制起始点定位和表面等离子体共振已经被开发来识别DNAORIs[5]。Lee和Bell[6]报道,与ORIs结合的起源识别复合物(ORC)可以被准确地检测到[7]。酿酒酵母的ORIs具有两种特征序列模式[8]:(1)ORIs具有高度特异性的序列模式,其中ORC识别t富17-bpARS基序及其共识序列,可能与邻近的B1/B2/B3元件[9]和(2)ORIs的核小体排斥[10]。然而,由于它们的时间和成本无效,建立了基于各种倾斜类型的计算工具。基于高通量测序的标记频率分析被用于分析原核生物[11]和真核生物[12]的复制特征和绘制ORIs。Ori-Finder[13]和Ori-Finder2[14]应用z曲线法和比较基因组学分析,对细菌和古细菌基因组中的ORCs进行了鉴定。其他的计算工具包括CG软件[15]和GraphDNA[16]。使用这种计算工具的局限性是,这些预测器仅基于包含ORIs信息的阳性样本构建。然而,在近十年来,各种计算方法的引入克服了这一限制,这些方法利用阳性和阴性样本来训练复制起源预测因子,并减少假阳性和假阴性的发生。Chen等人[17]预测了一种基于DNA结构特性来识别ORIs的方法,而Li等人[18]利用伪k元组核苷酸组成(PseKNC)来预测酿酒酵母的复制起始位点。随后,Zhang等人[19]和Xiao等[20]分别整合了二核苷酸的理化特征和位置特异性特征,采用一般的伪氨基酸组成[21],并提高了它们各自的预测因子的性能精度。后来,Liu等人[22]开发了一种名为“iRO-3wPseKNC”的预测因子,使用基于三窗口的PseKNC来预测四种酵母菌的ORIs。随后,同一组实施了不同长度的ORIs和鸟嘌呤/胞嘧啶(GC)不对称偏倚,并为两种酵母菌[23]开发了一种名为“iRO-PsekGCC”的预测算法。两个研究小组开发了他们各自的预测因子来预测酿酒酵母中的ORIs,使用相同的数据集,但不同的方法[24,25]。最近,Dao等人[26]提出了一个计算平台来识别来自真核生物的复制起源,
他们将特征选择技术应用于混合特征(Kmer和二进制编码的结合),并确定最优特征集,提高了对多个物种的预测精度。在现有的方法中,有四种方法适用于酵母菌的种类。其中,两种方法(iRO-3wPseKNC和iRO-PsekGCC)使用不同的ORI长度开发,其余两种方法使用固定的ORI长度开发。在这项工作中,我们的主要目标是从包含不同ORI长度的数据集中开发物种特异性的预测模型。尽管现有的方法(iRO-3wPseKNC和iRO-PsekGCC)的优点和性能改进的[22,23],不能排除某些限制:(i)现有方法仅采用单一编码和分类器;(ii)只有有限的特征工程努力,以探索更好的效率,因为现有工具只关注提取少数特征编码,(iii)未能评估模型的可转移性
通过解决上述问题,我们开发了一种新的物种特异性预测因子yORIpred,它可以准确预测酿酒酵母、乳酸酵母、毕赤酵母和裂殖酵母的裂ORIs。为了开发yORIpred,首先,通过探索5种不同的分类算法[随机森林(RF)、梯度增强(GB)、人工神经网络(ANN)、支持向量机(SVM)和极度随机树(ERT)]和8种不同的序列编码方案[Kmer组成、k间隔核酸对组成(CKSNAP)、电子-离子相互作用假势(EIIP)、二核苷酸理化性质(DPCP)、PseKNC、伪二核苷酸组成(PseDNC)、序列相关伪三核苷酸组成(SCPseTNC)和三核苷酸理化性质(TPCP)),为模型训练提供全面的特征信息。
值得注意的是,在基线模型生成过程中采用了两步特征选择方法。其次,将40个基线模型的ORIs预测概率视为新特征,采用迭代特征学习方法学习概率特征,以高度监督的迭代方式提高特征表示能力。我们的系统分析表明,与其他4个分类器相比,SVM学习到的特征表示在ORIs和非ORIs之间具有较高的区分分布特征。YORIpred在交叉验证(CV)分析和独立评估方面都显示出了良好的结果,从而表明迭代特征表示是其准确和稳健预测的唯一原因。我们预计,我们提出的预测器可能有助于识别新的ORIs,并有助于阐明其功能机制。
方法
1、数据集组成
最近,Liu等[22]基于DeOri数据库[14]构建了四种酵母(S. cerevisiae, K. lactis P.pastoris and S. pombe)不同长度的训练数据集。其中,S. cerevisiae包含341条ORI序列和343条非ORI序列;S. pombe包含339条ORI序列和336条非ORI序列;P. pastoris含306条ORI序列和303条非ORI序列,K. lactis包含148条ORI序列和148条非ORI序列由于DeOri数据库没有更新,本研究考虑了Liu等人的数据集,原因如下:他们应用了几种过滤方案,构建了一个可靠的训练数据集;(ii)没有序列从对于每个特定物种的数据集,与其他序列具有超过80%的序列一致性,(iii)使用这些数据集来开发模型,可以在现有方法和我们提出的方法之间进行公平的比较。
为了评估我们的预测模型,我们利用最近报道的ORI数据集(Ori-Finder3)[27]构建了一个独立的酿酒酵母物种数据集。一般来说,随机序列被用作非ORI,但我们使用的不是酿酒酵母的ORI功能以外的序列。具体来说,我们考虑了从iRSpot-Pse6NC2.0[28]下载的重组热点/冷点序列,并将它们视为非oris。随后,排除与训练数据集具有75%序列一致性的样本,得到67个ORIs和837个非ORIs。
特征编码
一般情况下,可变长度的DNA序列应通过特征提取[29]转换为固定长度的数值向量。在这里,我们使用了8个不同的特征编码,并评估了它们在从非ori序列中分类ori时的贡献,当将它们作为不同的机器学习(ML)分类器的输入时。简而言之,对每个描述符的描述如下:
基准分类器
采用SVM、ANN、RF、GB和ERT五种不同的分类器。由于SVM在yORIpred实现中的贡献非常重要,因此下面将详细介绍SVM及其使用情况。
SVM
SVM是一种功能强大的ML算法,已广泛应用于各种预测问题[39–45]。SVM的目的是将输入特征转换为一个高维空间,并找到能够最大化ORIs和非ORIs[46]之间的距离的最优超平面。在训练过程中,需要优化两个SVM参数,即核参数γ和惩罚参数C,以达到最佳性能。我们采用网格搜索方法对这些参数进行优化,搜索范围如下:2-15≤γ≤2−5,步长为−2,2-5≤C≤215,步长为2。值得一提的是,我们的初步分析表明,径向基函数比其他三个核函数(sigmoid、多项式和线性)具有更好的性能。在之前的研究[47–49]中,我们对剩余的分类器进行了简要的描述,表S1提供了其各自的分类器调优超参数搜索范围。值得注意的是,10倍CV被用于调整训练数据集上的ML参数,并检查了它们的性能。
迭代特征学习
我们采用迭代特征表示学习来开发一个鲁棒的预测模型。该方法已广泛应用于计算生物学[32,50]。图1显示了迭代特征学习框架,其中包括三个子步骤:(1)特征优化;(2)概率特征生成和(3)迭代特征生成,具体如下:
特征优化
一般情况下,原始特征维可能包含不相关信息,导致预测模型性能较差,模型鲁棒性较低。特征选择协议是开发预测模型,以减少或消除冗余,以提高模型性能和减少计算时间的重要步骤之一。为了包含每个描述符的更多相关信息,我们对7个特征编码(Kmer、CKSNAP、PseKNC、SCPseKNC、EIIP、DPCP和TPCP)进行了特征优化,并由于其特征维度较小而排除了PseDNC(18D)。采用系统的两步特征选择方法从原始特征维数中识别出最优特征集,其中第一步是对特征进行排序,然后进行序列前向搜索(SFS)。一般来说,极端GB(XGBoost)被称为一种预测算法[54]。除了预测外,它还可以有效地根据重要性评分对特征进行排序,并被广泛应用于各种问题中识别相关特征集[55,56]的方法。在这里,我们使用XGBoost对特征进行排序,并根据分数对它们按降序排序。随后,应用SFS从每个编码子集中识别最优特征子集。在SFS中,每次将k(k=2)特征添加到5个不同的ML分类器中,并使用10倍CV评估其性能。最终,在精度(ACC)被认为是每个分类器的最优模型。
概率特征的生成
从步骤1中,我们得到了40个最优模型(8个编码×5个分类器),它们的ORIs的预测概率被连接起来,并被认为是一个40D的新特征向量。
迭代特征生成
受深度神经网络[57]中分层学习特征的方法的启发,我们采用了类似的策略来开发最终的预测模型。在这里,我们分别使用了五种不同的分类器来进行迭代方法。过程如下:在迭代策略的第一次运行中,从第2步得到的40D概率特征向量输入到ML分类器(如SVM),并使用10倍CV建立相应的模型。从最优模型中得到的ORIs的预测概率,被认为是一个新的特征向量,与之前的40D向量相结合,得到41D特征向量。我们以迭代的方式重复这个过程,通过连续每一步训练多维特征,然后融合输出和输入特征,作为输入特征,得到ORIs的一维预测概率。这种迭代方法将在11轮后终止,以避免过拟合
特征融合法
特征融合方法在生物信息学和计算生物学的[58]中非常流行。除了迭代特征表示外,我们还采用了特征融合方法,并评估了该方法是否提高了预测性能。简而言之,将Kmer、CKSNAP、PseKNC、PseDNC、SCPseKNC、EIIP、DPCP和TPCP等8个不同的基于序列的编码载体合并如下:
FF是产生3597个D特征的顺序融合。值得注意的是,融合的特性可能包含直接影响性能的不相关的或相互作用的信息。因此,我们在FF上采用了两步特征选择方法(如上所述),分别采用五种不同的分类器,并建立了相应的最优模型。
评价指标
为了量化所开发模型的性能并对其中进行评估,我们采用了5个常用的评价指标[59–61]:敏感性(Sn)、特异性(Sp)、ACC、马修斯相关系数(MCC)、平衡准确性(BACC)。每个度量的定义表示为:
结果与讨论
评估不同的特征表示和分类器的表现
我们采用了5种不同的分类器(ANN、RF、GB、ERT和SVM)和8种不同的特征编码器(Kmer、CKSNAP、DPCP、PseDNC、TPCP、PseKNC、SCPseKNC和EIIP)来评估它们在每种酵母物种的ORIs预测中的贡献。
传统上,每个编码都被输入到5个不同的分类器中,并使用10倍CV建立了它们各自的预测模型。每个物种共开发了40个预测模型(8个编码×5ML分类器),其性能如图2和表S2-S5所示。图2显示,每个分类器的性能在不同的特征编码下都很幸运,其中它与其他分类器共享相似的MCC,有时MCC略高,而MCC略低。例如,使用CKSNAP、Kmer和TPCP的MCC基于SVM的表现突出,类似于使用DPCP、EIIP和SCPseTNC的不同分类器的MCC,而使用PseDNC和PseKNC则略有恶化(图2B)。与此同时,在其他三个物种(乳酸酵母、酿酒酵母和pombe)中也观察到了类似的现象。值得一提的是,我们没有观察到任何突出的分类器,无论使用八种不同的特征编码,都始终保持良好的表现。
为了获得每个分类器的概述性能,我们计算了8个编码的平均性能,如图S1所示。结果表明,RF、ERT和GB在0.690–0.700范围内与MCC的性能相似,优于SVM和ANN。在酿酒酵母中,RF、ERT和SVM在0.414–0.418范围内取得了与MCC相似的性能,优于GB和ANN。此外,SVM在其余两个数据集中都表现出了优越的性能。总体而言,以上分析表明,无论数据集如何,三个分类器(RF、ERT和SVM)的性能都是相似的,但略优于ANN和GB。
通过两步特征选择得到的特征优化结果
如上所述,我们获得了40个基线模型,只考虑了35个模型进行特征优化,由于特征维数较低(18D),排除了5个模型(基于PseDNC的5个分类器)。首先,我们使用XGBoost分别对7个编码的特征重要性得分进行预测,并选择非零重要性得分特征。图3A显示,特征编码具有小于100个原始维度(CKSNAP、EIIP和SCPseTNC),无论数据集如何,都具有86-100%的非零特征重要性得分。有趣的是,具有高维的特征(Kmer、DPCP、PseKNC和TPCP)包含包含非零特征值的显著减少特征。随后,我们根据这些非零特征的得分对其进行排序,并进行SFS分析,以确定最优特征集。

基于10倍交叉验证的四种酵母菌中,使用8种不同特征类型训练的不同分类器的性能比较。

.特征选择和优化的效果。(A)XGBoost使用4个物种特定数据集对7个不同编码识别出的重要特征的百分比。(B)最优特征集模型与控制模型之间的精度提高百分比。我们只考虑了特征选择和改进的模型,并从7个编码中排除了恶化的模型。图中提到了改进后的模型的数量。

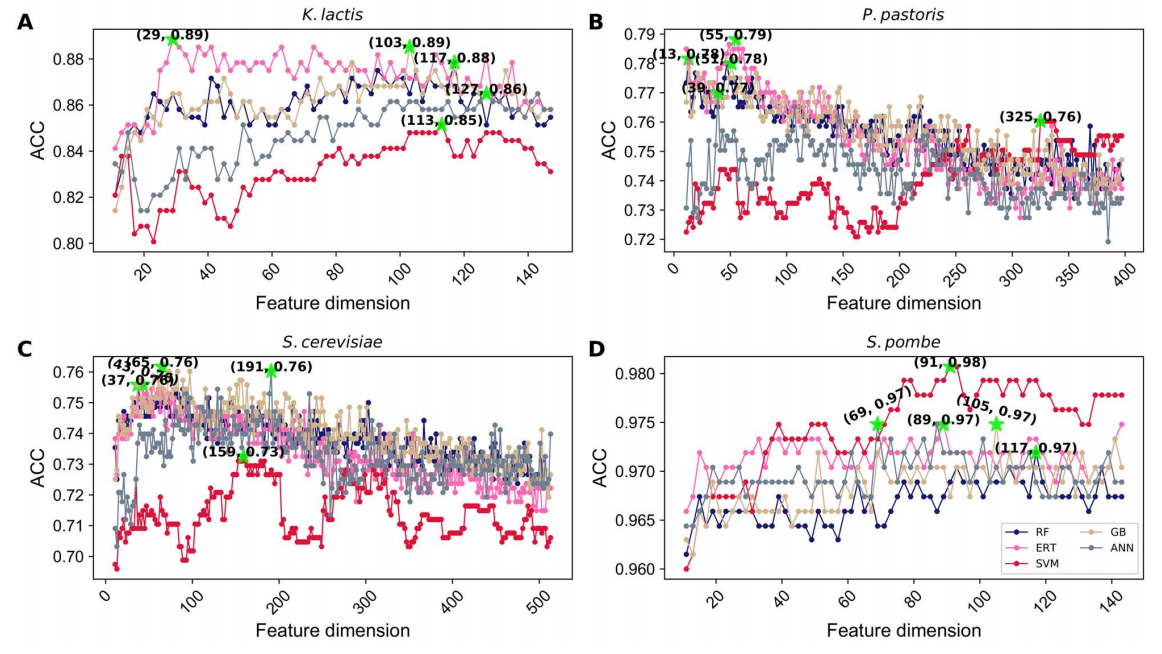
图4。在迭代特征学习过程中,五种不同分类器的性能比较。(A)性能-基于乳杆菌,最佳性能用绿色星号表示,5个分类器的最佳模型的比较用(B).表示©基于巴斯德氏的性能和5个分类器的最佳模型的比较见(D).(E)基于酿酒酵母的性能和来自5个分类器的最佳模型的比较如(F)所示。(G)基于S.pombe的性能和来自5个分类器的最佳模型的比较如(H)所示。
迭代特征学习过程中不同分类器的影响
我们从40个基线模型中获得了ORI的预测概率,并考虑了输入到5个不同分类器的新特征,并独立地执行了一个迭代特征学习过程。为了直观地检查结果,我们绘制了ACC作为增加了每个迭代步骤中特征维度的变化(图4)。结果表明,RF、ERT、GB和ANN具有共同的变化趋势,性能保持平衡(4A、C、E、G)。然而,SVM对两种植物(乳杆菌和巴斯德杆菌)具有普遍的发展趋势,ACC在开始时达到峰值,并趋于稳态(4a和C)。在酿酒酵母的情况下,SVM的ACC曲线最初增加,达到峰值并组装到稳定状态。接下来,我们比较了三个物种(乳酸菌、巴斯德菌和酿酒酵母)的最佳模型性能。图4B、D和F显示,SVM在所有五个指标上都显著优于其他分类器。在S.pombe的情况下,所有的分类器在第一次或第二次迭代中都达到了最大的ACC,其性能相似(图4H)。由于基于SVM的模型在我们的迭代方法中具有良好的性能和一致的性能,我们选择了它作为每个物种的最终模型,并将其命名为yORIpred。
基于SVM的迭代特征表示学习策略与同类相比显著提高了性能。然而,目前尚不清楚是简单的分类器选择起着关键作用还是其他隐藏因素。因此,我们对三个物种为了理解这个事件:(1)SVM基于其他分类器的概率特征来评估性能。SVM的性能拓扑被认为与4个分类器相似(数据未显示),表明4个分类器生成的特征(使用迭代方法)在ORIs和非ORIs之间表现出中等的区分能力。将基于(2)SVM的概率特征输入到四个分类器中,并在每次迭代中评估它们的性能。从图5可以看出,ACC四种分类器的拓扑都与SVM相同,这表明每次迭代时基于SVM生成的预测概率值在ORIs和非ORIs之间具有非常高的区分能力。因此,不仅是SVM,而且其他方法也表现得非常好。总的来说,上述分析表明,分类器对生成概率特征的选择(SVM)(在迭代方法中)对yoripred的性能起着重要作用。
yORIpred与特征融合方法的性能比较
近年来,一些研究证明了特征融合方法在一些预测问题[56,63]中的优势。因此,我们应用特征融合方法来研究与迭代特征学习方法相比,它是否能提高预测性能。准确地说,所有8种不同的编码都线性组合,生成了一个3597D特征向量。对于每个物种,我们首先对XGBoost识别出的非零分数的特征进行排序,然后使用5种不同的分类器(如上所述)应用SFS,识别出相应的最优模型。图6显示了每个物种的五种不同分类器的性能。有趣的是,三种分类器(RF,ERT和GB)的ACC曲线拓扑结构是相似的,无论物种如何。 ANN和SVM具有不同的ACC曲线拓扑结构。最优特征集所获得的最大ACC用绿色星号表示。接下来,我们将特征融合得到的最优模型与yORIpred进行了比较,结果如表1所示,其中特征融合模型以前缀FF命名。结果表明,yORIpred的性能始终优于特征融合模型。为了得到yORIpred和其他方法之间的统计学意义,我们比较了两个AUC值,并使用双尾检验[62]计算P值。通过应用P值为0.05的阈值,yORIpred显著优于两个物种(巴斯德酵母和酿酒酵母)的所有特征融合模型。在K.lactis中,yORIpred的表现优于FF-RF、FF-ERT和FF-GB,且显著优于其他两种模型。此外,yORIpred的性能与S.pombe中的其他FF模型相似。总之,上述的比较分析强调,迭代学习方法是yoripred提高性能的唯一原因。
从特征融合中选择五种不同分类器的最优特征集模型。预测精度与特征子集维数的关系曲线。图中的曲线反映了特征子集维度对预测精度的变化。每个分类器中选择的模型在绿色星号中提到。
yORIpred与现有预测器在同一训练数据集上的性能比较
我们比较了yORIpred与现有方法的性能,即iRO-3wPseKNC和iRO-PsekGCC,其中两种方法都是使用相同的训练数据集开发的,分别包含4个和2个物种特异性预测模型。通常,使用相同的数据集比较CV或独立性能是一种更客观的方法。表2显示了yORIpred与现有预测因子之间的性能比较结果,显示yORIpred在MCC、ACC、Sn、Sp和AUC方面比其他预测因子具有更好的预测性能。其中,yORIpred获得的MCC比iRO-3wPseKNC高16.2%;31.0–37.0%高于两种现有方法;22.5–29.6%高于目前两种酿酒酵母方法,22.5–29.6%比iRO-3wPseKNC高4.0%。通过0.05的统计临界值,yORIpred对乳酸杆菌、酿酒酵母的影响显著优于iRO-3wPseKNC,对pombe的影响略优于iRO-3wPseKNC。此外,yORIpred对两种物种均显著优于iRO-PseKGCC。需要注意的是,在现有方法中使用的相同的射频分类器具有不同的特征编码。因此,它不能捕捉到能够很好地区分ori和非ori的模式。然而,我们的方法克服了现有方法和本研究中采用的特征融合方法的局限性,表现出了良好的性能。出现的可能原因优越的性能包括:(i)整合不同的分类器和特征编码方案,生成概率特征;(ii)在迭代特征学习和选择SVM过程中对不同的分类器进行系统评价;(iii)SVM生成的概率特征显示出ORIs对非ORIs具有很高的识别能力。
对独立数据集的性能验证
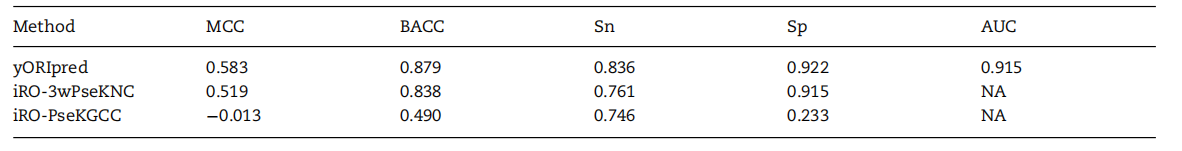
我们比较了yORIpred与现有方法的预测性能,该数据集包括67个ORIs和837个非ORIs。由于数据集是不平衡的,使用ACC进行比较并不简单;因此,我们使用BACC对方法进行排序,如表3所示。对比结果显示,yORIpred的MCC和BACC分别为0.583和0.879。其中,用yORIpred进行的MCC比第二优的方法iRO-3wPseKNC高6.4%。在现有的方法中,iRO-PseKGCC在我们的评价中性能较低。总体来说,yORIpred不仅在训练数据集上表现良好,而且在独立评估中复制了其CV性能,从而突出了其在ORIs预测中的稳定性和鲁棒性。
网站服务开发
作为已开发的yORIpred方法的实现,我们已经在http://thegleelab.org/yORIpred上提供了yORIpred的在线网络服务器版本。该web服务器配备了16核,64GB内存和一个2TB的硬盘。为了利用yORIpredweb服务器,用户应该从主页中选择特定的物种,并在文本框中输入DNA序列,或通过文件选择对话框以快速自适应收缩阈值算法(FASTA)格式上传序列。预测结果以表格格式提供,详细说明了序列号、FASTAID、预测类(ORIs或非ORIs)和ORIs的预测概率。值得注意的是,ORI的概率分数在0到1的范围内,其中概率分数接近1表示结果可能是ORI,分数接近0表示结果不太可能是ORI。关于使用yORIpredweb服务器的更详细说明可以在web服务器的自述页面中找到。
yORIpred与基于特征融合方法的预测器的性能比较

yORIpred与现有方法在同一训练数据集上的性能比较

各种方法在酿酒酵母独立数据集上的性能比较
最后
以上就是追寻汉堡最近收集整理的关于论文解读《通过迭代特征表示计算预测物种特异性酵母DNA复制起源》摘要介绍方法结果与讨论的全部内容,更多相关论文解读《通过迭代特征表示计算预测物种特异性酵母DNA复制起源》摘要介绍方法结果与讨论内容请搜索靠谱客的其他文章。








发表评论 取消回复