欢迎关注”生信修炼手册”!
对于单细胞转录组的数据,常用的降维方法有以下3种
PCA
t-SNE
Difffusion map
通过scater这个R包,可以方便的进行降维分析,安装方式如下
BiocManager::install("scater", version = "3.8")具体的操作步骤如下
1. 构建SingleCellExperiment对象
对于单细胞的数据,专门制定了一个名为SingleCellExperiment的类,用来存储相关数据。
我们首先要做的就将相关数据导入到R中,只需要下两种数据即可,第一种是基因的表达量数据,每一行代表一个基因,每一列代表一个细胞,示意如下
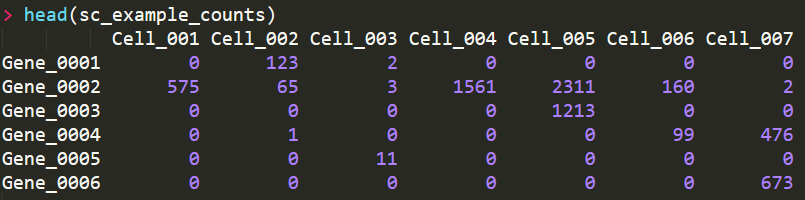
第二种是细胞的相关信息,可以是细胞的名字,采样时间,来源组织,处理条件等metadata, 每一行是一个细胞,每一列是一种属性,示意如下
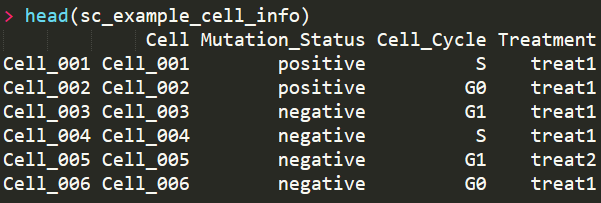
通过这两种数据,就可以构建出一个SingleCellExperiment对象,代码如下
sce <- SingleCellExperiment(
assays = list(counts = sc_example_counts),
colData = sc_example_cell_info
)
# 归一化
sce <- normalize(sce)注意必须要进行归一化操作。
2. PCA
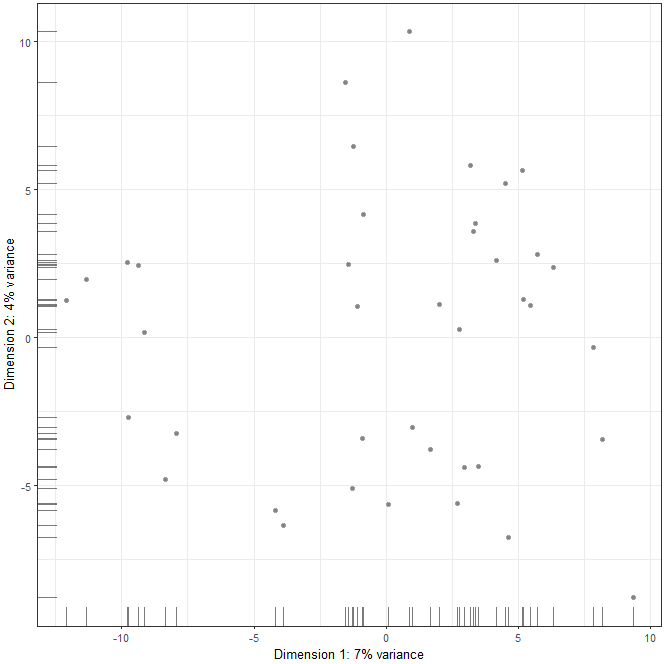
PCA是应用的最广泛的降维方法,在scater中,通过一下方式可以快速的得到PCA降维后的结果,代码如下
plotPCA(sce)生成的图片如下
2. t-SNE
t-SNE降维算法的代码如下
set.seed(1000)
sce <- runTSNE(
sce,
perplexity = 10,
use_dimred = "PCA",
n_dimred = 10)
# 画图
plotTSNE(sce, colour_by="Treatment")生成的图片如下

本质上是通过调用Rtsne这个包来进行t-SNE降维分析。
3. Diffusion Map
Diffusion Map简称DM降维算法,代码如下
sce <- runDiffusionMap(sce)
plotDiffusionMap(sce)生成的图片如下
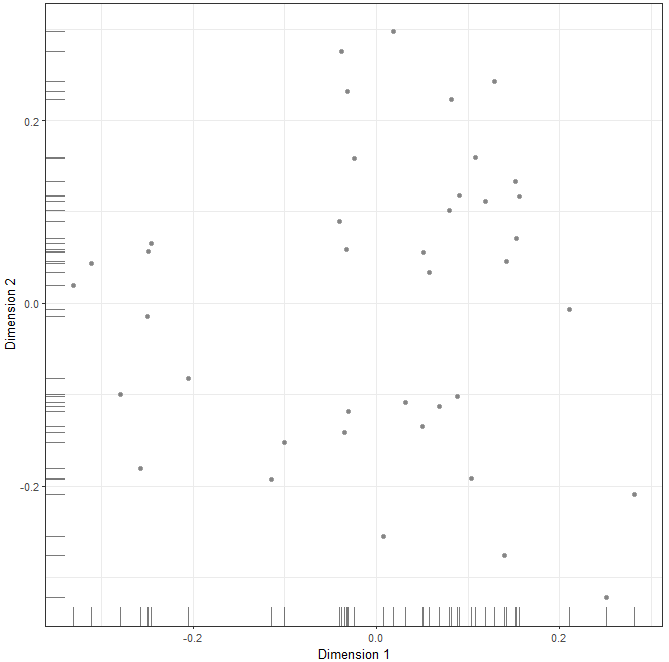
本质上是通过调用destiny这个包来进行降维分析。
scater这个R包不仅提供了各种降维分析的算法,还提供了数据QC, 基因表达量可视化等功能,更多用法请参阅官方文档。
·end·
—如果喜欢,快分享给你的朋友们吧—
扫描关注微信号,更多精彩内容等着你!

最后
以上就是愉快微笑最近收集整理的关于使用scater包对单细胞转录组数据进行降维分析的全部内容,更多相关使用scater包对单细胞转录组数据进行降维分析内容请搜索靠谱客的其他文章。
本图文内容来源于网友提供,作为学习参考使用,或来自网络收集整理,版权属于原作者所有。








发表评论 取消回复