【R语言数据科学】
- ????个人主页:JOJO数据科学
- ????个人介绍:统计学top3高校统计学硕士在读
- ????如果文章对你有帮助,欢迎✌
关注、????点赞、✌收藏、????订阅专栏- ✨本文收录于【R语言数据科学】本系列主要介绍R语言在数据科学领域的应用包括:
R语言编程基础、R语言可视化、R语言进行数据操作、R语言建模、R语言机器学习算法实现、R语言统计理论方法实现。本系列会坚持完成下去,请大家多多关注点赞支持,一起学习~,尽量坚持每周持续更新,欢迎大家订阅交流学习!

文章目录
- 【R语言数据科学】
- 1.preProcess 函数
- 2.标准化处理
- 3.转换预测变量
- 4.同时变换
- 5.计算类距离
1.preProcess 函数
preProcess 类可用于预测变量的许多操作,包括center和scale。函数 preProcess 估计每个操作所需的参数, predict.preProcess 用于将它们应用于特定的数据集。该函数也可以是调用train函数时的接口。
在接下来的几节中将描述几种类型的技术,然后使用另一个示例来演示如何使用多种方法。请注意,在所有情况下,preProcess 函数都会从特定数据集(例如训练集)估计它需要的任何内容,然后将这些转换应用于任何数据集,而无需重新计算值
2.标准化处理
在下面的示例中,一半的 MDrr 数据用于估计预测变量的位置和规模。函数 preProcess 实际上并不预处理数据。 predict.preProcess 用于预处理这个和其他数据集。
set.seed(96)
inTrain <- sample(seq(along = mdrrClass), length(mdrrClass)/2)
training <- filteredDescr[inTrain,]
test <- filteredDescr[-inTrain,]
trainMDrr <- mdrrClass[inTrain]
testMDrr <- mdrrClass[-inTrain]
preProcValues <- preProcess(training, method = c("center", "scale"),range=c(0,1))
trainTransformed <- predict(preProcValues, training)
testTransformed <- predict(preProcValues, test)
preProcess 选项“范围”将数据缩放到零和一之间的间隔。
preProcess 可用于仅基于训练集中的信息来估算数据集。一种方法是使用 K 最近邻。对于任意样本,在训练集中找到 K 个最近邻,并使用这些值(例如使用平均值)估算预测变量的值。使用此方法将自动触发 preProcess 以居中和缩放数据,而不管方法参数中的内容是什么。
3.转换预测变量
在某些情况下,需要使用主成分分析 (PCA) 将数据转换到一个较小的子空间,其中新变量彼此不相关。 preProcess 类可以通过在方法参数中包含“pca”来应用此转换。这样做也将强制对预测变量进行缩放。请注意,当请求 PCA 时,predict.preProcess 会将列名更改为 PC1、PC2 等。
类似地,独立分量分析 (ICA) 也可用于查找新变量,这些变量是原始集合的线性组合,使得分量是独立的(与 PCA 中的不相关相反)。新变量将被标记为 IC1、IC2 等。
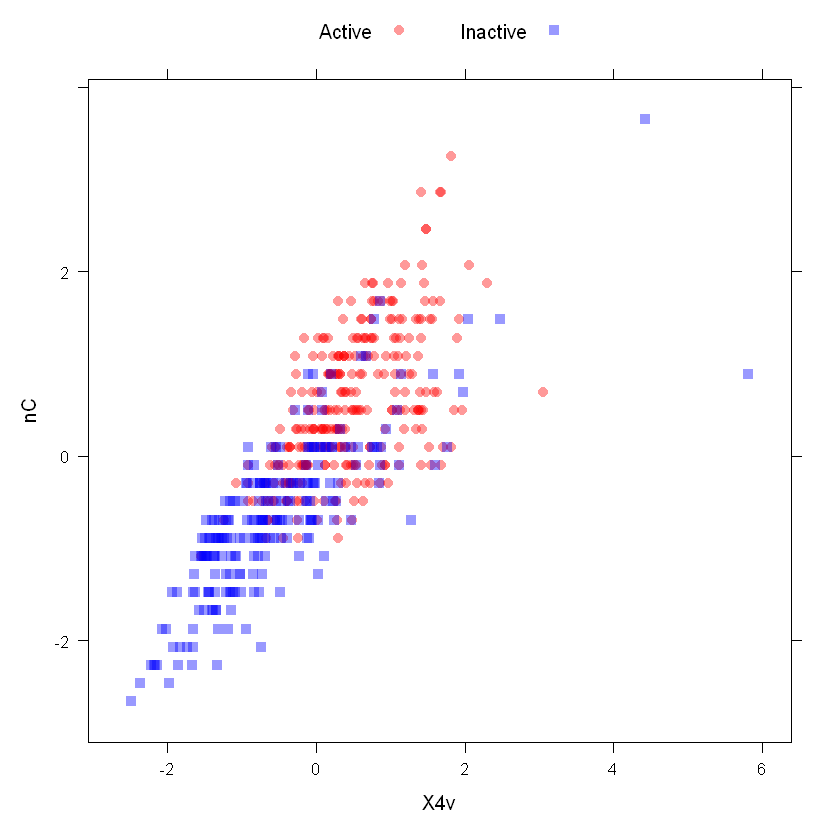
“空间符号”变换 (Serneels et al, 2006) 将预测变量的数据投影到 p 维的单位圆上,其中 p 是预测变量的数量。本质上,数据向量除以其范数。下面的两个图显示了空间符号转换前后 MDrr 数据中的两个居中和缩放的描述符。在应用此转换之前,应将预测变量居中并按比例缩放。
library(AppliedPredictiveModeling)
transparentTheme(trans = .4)
plotSubset <- data.frame(scale(mdrrDescr[, c("nC", "X4v")]))
xyplot(nC ~ X4v,
data = plotSubset,
groups = mdrrClass,
auto.key = list(columns = 2))
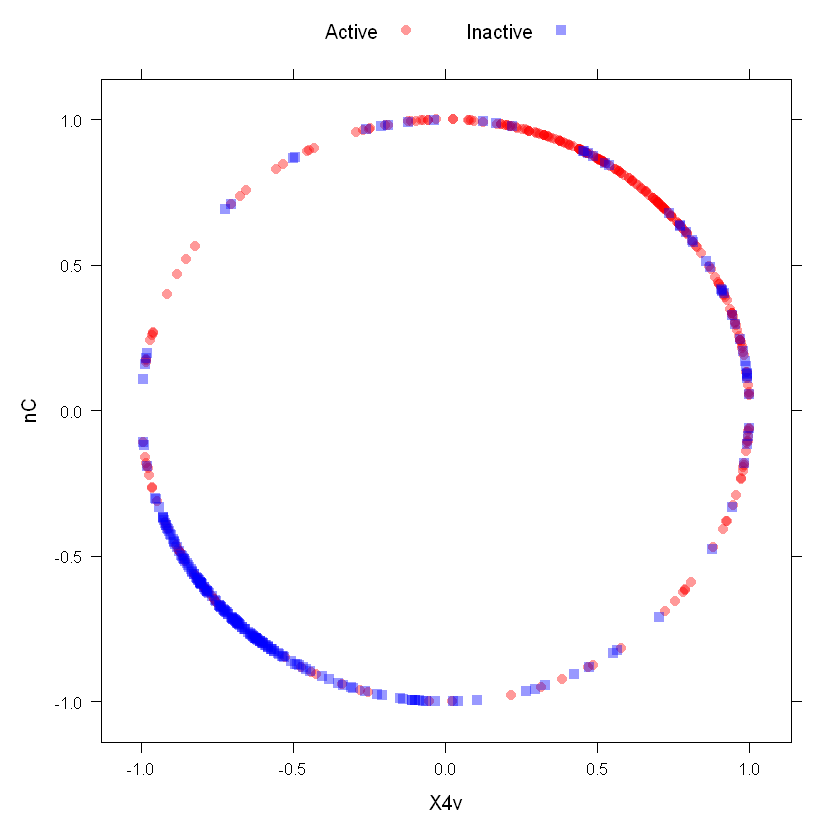
使用spatial sign之后
transformed <- spatialSign(plotSubset)
transformed <- as.data.frame(transformed)
xyplot(nC ~ X4v,
data = transformed,
groups = mdrrClass,
auto.key = list(columns = 2))
如果数据大于零,另一个选项“BoxCox”将估计预测变量的 Box–Cox 变换。
Box-Cox变换是Box和Cox在1964年提出的一种广义幂变换方法,是统计建模中常用的一种数据变换,用于连续的响应变量不满足正态分布的情况。Box-Cox变换之后,可以一定程度上减小不可观测的误差和预测变量的相关性。
Cox变换的主要特点是引入一个参数lambda,通过数据本身估计该参数进而确定应采取的数据变换形式,Box-Cox变换可以明显地改善数据的正态性、对称性和方差相等性,又不丢失信息,后经过一定的推广和改进,扩展了其应用范围。
y
(
λ
)
=
y
λ
−
1
λ
λ
≠
0
l
o
g
(
y
)
,
l
a
m
b
d
a
=
0
y(lambda) = frac{y^{lambda}-1}{lambda} lambda neq 0 log(y),lambda = 0
y(λ)=λyλ−1λ=0log(y),lambda=0
Box Cox变换的核心参数是lambda(λ),其范围从-5到5。所以我们主要目的在于通过一定的方法,选择除最佳的lambda值。
preProcValues2 <- preProcess(training, method = "BoxCox")
trainBC <- predict(preProcValues2, training)
testBC <- predict(preProcValues2, test)
preProcValues2
Created from 264 samples and 31 variables
Pre-processing:
- Box-Cox transformation (31)
- ignored (0)
Lambda estimates for Box-Cox transformation:
Min. 1st Qu. Median Mean 3rd Qu. Max.
-2.0000 -0.2500 0.5000 0.4387 2.0000 2.0000
NA 值对应于无法转换的预测变量。此转换要求数据大于零。两个类似的变换,Yeo-Johnson 和 Manly (1976) 的指数变换也可用于 preProcess。
4.同时变换
在应用预测建模中有一个案例研究,其中预测了高性能计算环境中作业的执行时间。数据如下:
library(AppliedPredictiveModeling)
data(schedulingData)
str(schedulingData)
'data.frame': 4331 obs. of 8 variables:
$ Protocol : Factor w/ 14 levels "A","C","D","E",..: 4 4 4 4 4 4 4 4 4 4 ...
$ Compounds : num 997 97 101 93 100 100 105 98 101 95 ...
$ InputFields: num 137 103 75 76 82 82 88 95 91 92 ...
$ Iterations : num 20 20 10 20 20 20 20 20 20 20 ...
$ NumPending : num 0 0 0 0 0 0 0 0 0 0 ...
$ Hour : num 14 13.8 13.8 10.1 10.4 ...
$ Day : Factor w/ 7 levels "Mon","Tue","Wed",..: 2 2 4 5 5 3 5 5 5 3 ...
$ Class : Factor w/ 4 levels "VF","F","M","L": 2 1 1 1 1 1 1 1 1 1 ...
数据是分类和数字预测变量的混合。假设我们想对连续预测变量使用 Yeo-Johnson 变换,然后对它们进行中心化和缩放。我们还假设我们将运行基于树的模型,因此我们可能希望将因子保留为因子(而不是创建虚拟变量)。我们在除最后一列之外的所有列上运行该函数
pp_hpc <- preProcess(schedulingData[, -8],
method = c("center", "scale", "YeoJohnson"))
pp_hpc
Created from 4331 samples and 7 variables
Pre-processing:
- centered (5)
- ignored (2)
- scaled (5)
- Yeo-Johnson transformation (5)
Lambda estimates for Yeo-Johnson transformation:
-0.08, -0.03, -1.05, -1.1, 1.44
transformed <- predict(pp_hpc, newdata = schedulingData[, -8])
head(transformed)
| Protocol | Compounds | InputFields | Iterations | NumPending | Hour | Day | |
|---|---|---|---|---|---|---|---|
| <fct> | <dbl> | <dbl> | <dbl> | <dbl> | <dbl> | <fct> | |
| 1 | E | 1.2289592 | -0.6324580 | -0.0615593 | -0.554123 | 0.004586516 | Tue |
| 2 | E | -0.6065826 | -0.8120473 | -0.0615593 | -0.554123 | -0.043733201 | Tue |
| 3 | E | -0.5719534 | -1.0131504 | -2.7894869 | -0.554123 | -0.034967177 | Thu |
| 4 | E | -0.6427737 | -1.0047277 | -0.0615593 | -0.554123 | -0.964170752 | Fri |
| 5 | E | -0.5804713 | -0.9564504 | -0.0615593 | -0.554123 | -0.902085020 | Fri |
| 6 | E | -0.5804713 | -0.9564504 | -0.0615593 | -0.554123 | 0.698108782 | Wed |
输出中标记为“忽略”的两个预测变量是两个因子预测变量。这些不会改变,但数字预测变量会被转换。但是,待处理作业数量的预测器具有非常稀疏且不平衡的分布:
mean(schedulingData$NumPending == 0)
0.756176402678365
对于其他一些模型,这可能是一个问题(特别是如果我们对数据进行重新采样或下采样)。在运行预处理计算之前,我们可以添加一个过滤器来检查零或接近零方差的预测变量:
pp_no_nzv <- preProcess(schedulingData[, -8],
method = c("center", "scale", "YeoJohnson", "nzv"))
pp_no_nzv
Created from 4331 samples and 7 variables
Pre-processing:
- centered (4)
- ignored (2)
- removed (1)
- scaled (4)
- Yeo-Johnson transformation (4)
Lambda estimates for Yeo-Johnson transformation:
-0.08, -0.03, -1.05, 1.44
predict(pp_no_nzv, newdata = schedulingData[1:6, -8])
| Protocol | Compounds | InputFields | Iterations | Hour | Day | |
|---|---|---|---|---|---|---|
| <fct> | <dbl> | <dbl> | <dbl> | <dbl> | <fct> | |
| 1 | E | 1.2289592 | -0.6324580 | -0.0615593 | 0.004586516 | Tue |
| 2 | E | -0.6065826 | -0.8120473 | -0.0615593 | -0.043733201 | Tue |
| 3 | E | -0.5719534 | -1.0131504 | -2.7894869 | -0.034967177 | Thu |
| 4 | E | -0.6427737 | -1.0047277 | -0.0615593 | -0.964170752 | Fri |
| 5 | E | -0.5804713 | -0.9564504 | -0.0615593 | -0.902085020 | Fri |
| 6 | E | -0.5804713 | -0.9564504 | -0.0615593 | 0.698108782 | Wed |
请注意,一个预测器被标记为“removed”,并且处理后的数据缺少稀疏预测器。
5.计算类距离
caret 包含根据到类质心的距离生成新预测变量的函数(类似于线性判别分析的工作原理)。对于因子变量的每个级别,都会计算类质心和协方差矩阵。对于新样本,计算到每个类质心的 Mahalanobis 距离,并可用作额外的预测器。当真正的决策边界实际上是线性的时,这对非线性模型很有帮助。
在类中的预测变量多于样本的情况下,classDist 函数有pca和keep参数允许在每个类中使用主成分分析来避免奇异协方差矩阵的问题。
predict.classDist 然后用于生成类距离。默认情况下,距离会被记录,但这可以通过 predict.classDist 的 trans 参数进行更改。
例如,我们可以使用 MDrr 数据。
centroids <- classDist(trainBC, trainMDrr)
distances <- predict(centroids, testBC)
distances <- as.data.frame(distances)
head(distances)
| dist.Active | dist.Inactive | |
|---|---|---|
| <dbl> | <dbl> | |
| ACEPrOMAZINE | 3.787139 | 3.941234 |
| ACEPrOMETAZINE | 4.306137 | 3.992772 |
| MESOrIDAZINE | 3.707296 | 4.324115 |
| PErIMETAZINE | 4.079938 | 4.117170 |
| PrOPErICIAZINE | 4.174101 | 4.430957 |
| DUOPErONE | 4.355328 | 6.000025 |
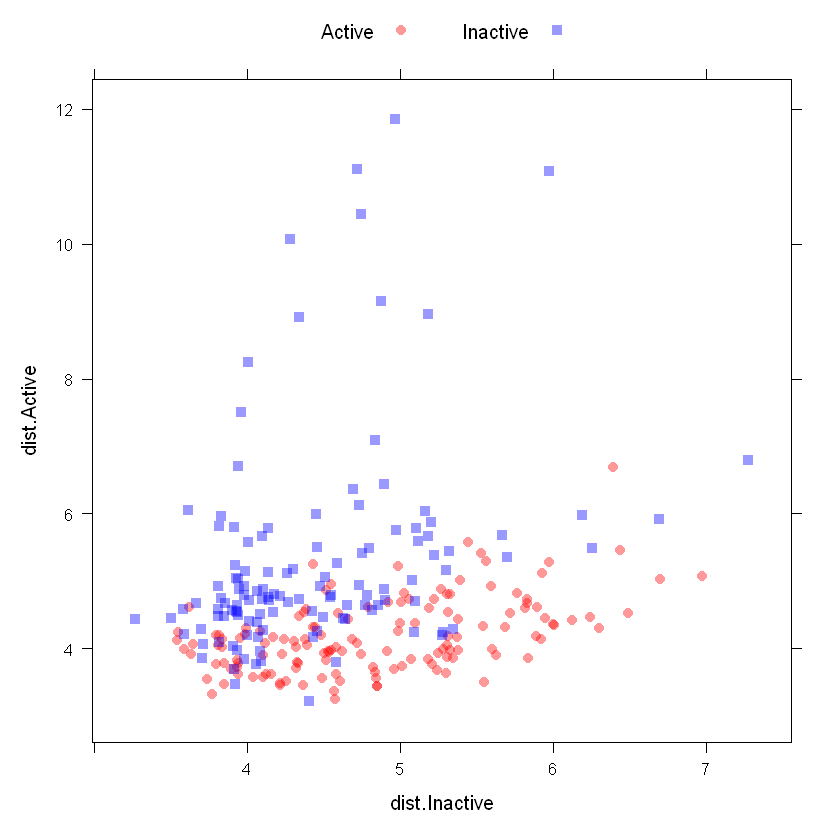
此图像显示了保留样本的类距离的散点图矩阵:
xyplot(dist.Active ~ dist.Inactive,
data = distances,
groups = testMDrr,
auto.key = list(columns = 2))
????本章的介绍到此介绍,如果文章对你有帮助,请多多点赞、收藏、评论、关注支持!!
最后
以上就是健壮花卷最近收集整理的关于R语言caret机器学习(三):数据预处理下集【R语言数据科学】1.preProcess 函数2.标准化处理3.转换预测变量4.同时变换5.计算类距离的全部内容,更多相关R语言caret机器学习(三):数据预处理下集【R语言数据科学】1.preProcess内容请搜索靠谱客的其他文章。








发表评论 取消回复