MRI to FDG-PET: Cross-Modal Synthesis Using 3D U-Net for Multi-modal Alzheimer’s Classification
- 方法
- 数据集
- 预处理
- 网络结构
- 实验
- 分类实验
这篇文章发表于2018年的International Workshop on Simulation and Synthesis in Medical Imaging,论文使用了一个3D U-Net结构从3D MRI中合成3D PET,在定量评估上,使用了MAE、PSNR与SSIM三种指标,并且将合成的PET与MRI一同用于AD分类中,相比只用MRI的70.18%分类精度,使用MRI+合成PET后的结果达到了74.43%。
方法
数据集
使用了来自ADNI数据集的384个同时具有MRI和PET影像的患者(其中192个正常,192个AD)。
预处理
首先,使用FSL软件将MR配准到MNI模板空间,它是标准模板。 随后对MR图像进行头盖骨剥离,并归一化到[0,1](避免梯度爆炸和收敛问题)。对PET图像,也配准到相同的MNI模板上,不进行头盖骨剥离,但归一化到[0,1]。值得注意的是,文章提到将处理好的MRI和PET图像使用FSL分割为灰质、白质、脑脊髓(只进行灰质部分的MRI-PET转换?)。并且将预处理好的MRI和PET影像都缩小到了 64 × 64 × 64 64times64times64 64×64×64大小。
网络结构
3D U-Net结构。目标是学习一个从MRI提取的灰质到PET提取的灰质的映射(但是结果又是整张图像的结果?)。损失函数为二值交叉熵损失。
实验
使用了9折交叉验证,1折用作测试集,1折用作验证集,剩下7折用于训练数据。训练了10个epoch。则应该是AD和NC混合训练,并没有单独对某一种训练一个模型。
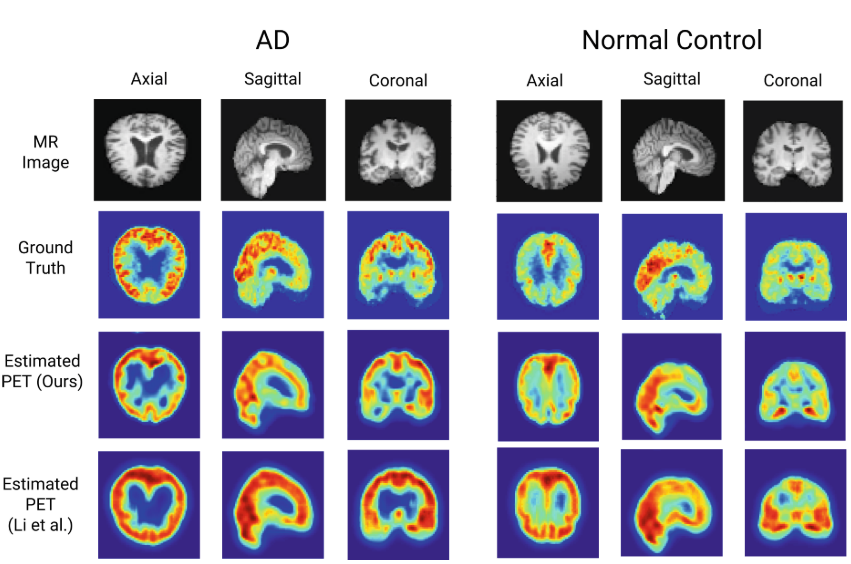
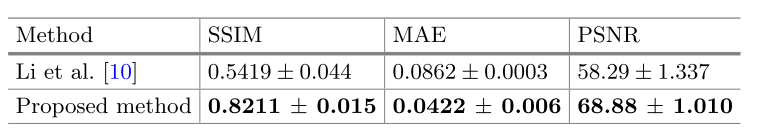
分类实验
使用了L2范数的Logistic regression(逻辑回归,一种经典的二分类算法,参考从原理到应用:简述Logistic回归算法)。并同样使用了9折交叉验证,作者进行的对比如下:采用原始的MRI数据得到一个分类结果、采用原始的PET数据得到一个结果、采用合成的PET数据得到一个结果、采用原始的MRI和合成的PET得到一个结果四者的对比,作者通过采用原始MRI+合成PET相比原始MRI的结果要高说明合成的PET是有意义的(这个对比很不科学?,不应该对比MRI+PET与MRI+合成的PET的结果吗?而且原始数据中的MRI不是已经有对应的PET了?),结果如下:
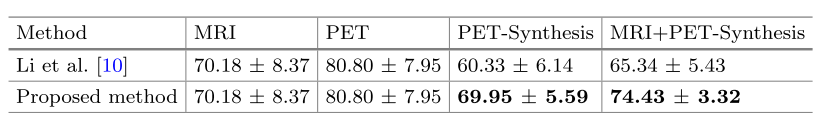
作者还进行了一个配对样本t检验(用于判断样本是否足以代表整体),检验结果显示使用MRI和合成PET图像时,分类准确性略有改善(
p
=
0.06
p=0.06
p=0.06)。
最后
以上就是粗犷背包最近收集整理的关于MRI to FDG-PET: Cross-Modal Synthesis Using 3D U-Net for Multi-modal Alzheimer’s Classification论文解读方法实验的全部内容,更多相关MRI内容请搜索靠谱客的其他文章。








发表评论 取消回复